၁။ တုံ့ပြန်မှုစနစ်၏ အာရုံခံနိုင်စွမ်းကို တိုးစေသည် ။
1. အရည်အသွေးမြင့် RNA ကို သီးခြားခွဲထုတ်ပါ
အောင်မြင်သော cDNA ပေါင်းစပ်မှုသည် အရည်အသွေးမြင့် RNA မှလာသည်။အရည်အသွေးမြင့် RNA သည် အနည်းဆုံး EDTA သို့မဟုတ် SDS ကဲ့သို့သော ပြောင်းပြန် transcriptase inhibitors များ ကင်းစင်သင့်သည်။RNA ၏ အရည်အသွေးသည် သင် cDNA သို့ ကူးယူနိုင်သော ဆက်တိုက်အချက်အလက် အများဆုံးပမာဏကို ဆုံးဖြတ်သည်။ယေဘူယျအားဖြင့် RNA သန့်စင်မှုနည်းလမ်းသည် guanidine isothiocyanate/acid phenol ကိုအသုံးပြုသည့် အဆင့်တစ်ဆင့်နည်းလမ်းဖြစ်သည်။RNase ၏ခြေရာခံပမာဏအားဖြင့် ညစ်ညမ်းခြင်းမှ ကာကွယ်ရန်၊ အထူးသဖြင့် ရေရှည်သိုလှောင်မှုအတွက် အရည်အသွေးမြင့် RNA ကို ထိန်းသိမ်းရန် RNase ကြွယ်ဝသောနမူနာများ (ဥပမာ ပန်ကရိယ) မှခွဲထုတ်ထားသော RNA ကို formaldehyde တွင် သိမ်းဆည်းထားရန် လိုအပ်ပါသည်။ကြွက်အသည်းမှ ထုတ်ယူရရှိသော RNA သည် ရေတွင် တစ်ပတ်ကြာ သိမ်းဆည်းပြီးနောက် အခြေခံအားဖြင့် ဆုတ်ယုတ်သွားပြီး ကြွက်သရက်ရွက်မှ ထုတ်ယူသော RNA သည် ရေတွင် ၃ နှစ်ကြာ သိမ်းဆည်းပြီးနောက် တည်ငြိမ်နေပါသည်။ထို့အပြင်၊ 4 kb ထက် ပိုရှည်သော စာသားများသည် သေးငယ်သော စာသားများထက် ခြေရာခံ RNases ဖြင့် ပြိုကွဲပျက်စီးခြင်းသို့ ပိုမိုထိခိုက်လွယ်ပါသည်။သိမ်းဆည်းထားသော RNA နမူနာများ၏ တည်ငြိမ်မှုကို တိုးမြှင့်ရန်၊ RNA ကို ဒိုင်းယွန်းပုံစံဖော်မိုင်းတွင် ပျော်ဝင်ပြီး -70°C တွင် သိမ်းဆည်းနိုင်သည်။RNA ကို ထိန်းသိမ်းရာတွင် အသုံးပြုသည့် Formamide သည် RNA ပြိုပျက်သွားသော အပျက်အစီးများ ကင်းစင်ရပါမည်။ပန်ကရိယမှ RNA ကို formamide တွင် အနည်းဆုံး တစ်နှစ်ကြာ ထိန်းသိမ်းထားနိုင်သည်။RNA ကိုအသုံးပြုရန်ပြင်ဆင်သောအခါတွင်၊ သင်သည် RNA ကိုရေခဲစေရန်အောက်ပါနည်းလမ်းကိုသုံးနိုင်သည်- NaCl ကို 0.2M နှင့် Ethanol ပမာဏ 4 ဆကိုထည့်ပါ၊ အခန်းအပူချိန်တွင် 3-5 မိနစ်ခန့်ထားကာ 10,000 × g တွင် 5 မိနစ်ကြာ centrifuge ။
2. RNaseH-inactive (RNaseH-) reverse transcriptase ကိုသုံးပါ
RNase inhibitors များကို cDNA ပေါင်းစပ်မှု၏ အရှည်နှင့် အထွက်နှုန်းကို တိုးမြင့်ရန်အတွက် ပြောင်းပြန် ကူးယူတုံ့ပြန်မှုများတွင် မကြာခဏ ပေါင်းထည့်ပါသည်။cDNA ပေါင်းစပ်ခြင်းမပြုမီ လုပ်ငန်းစဉ်သည် inhibitor ကို denature စေပြီး၊ ထို့ကြောင့် RNA ပြိုကွဲနိုင်သည့် ချည်နှောင်ထားသော RNase ကို စွန့်ထုတ်နိုင်သော ကြားခံနှင့် လျှော့ချအေးဂျင့် (ဥပမာ DTT ကဲ့သို့) တွင် RNase inhibitors များကို ပေါင်းထည့်သင့်သည်။ပရိုတိန်း RNase inhibitors များသည် RNase A, B, C အားဖြင့် RNA ၏ပြိုကွဲမှုကိုသာ ကာကွယ်ပေးပြီး အရေပြားပေါ်ရှိ RNase ကို မကာကွယ်နိုင်ပါ၊ ထို့ကြောင့် အဆိုပါ inhibitors များကို အသုံးပြုနေသော်လည်း သင့်လက်ချောင်းများမှ RNase ကို မမိတ်ဆက်မိစေရန် သတိထားပါ။
Reverse transcriptase သည် RNA မှ cDNA သို့ပြောင်းလဲခြင်းကို လှုံ့ဆော်ပေးသည်။M-MLV နှင့် AMV နှစ်မျိုးလုံးတွင် ၎င်းတို့၏ကိုယ်ပိုင် polymerase လုပ်ဆောင်မှုအပြင် endogenous RNaseH လုပ်ဆောင်ချက် ရှိသည်။RNaseH လုပ်ဆောင်ချက်နှင့် polymerase လုပ်ဆောင်ချက်တို့သည် RNA နမူနာပုံစံနှင့် DNA primer သို့မဟုတ် cDNA တိုးချဲ့ကြိုးကြားရှိ ပေါင်းစပ်ကြိုးမျှင်အတွက် အချင်းချင်း ပြိုင်ဆိုင်ကြပြီး RNA:DNA ရှုပ်ထွေးမှုတွင် RNA ကြိုးမျှင်ကို ကျဆင်းစေသည်။RNaseH လုပ်ဆောင်ချက်ဖြင့် ပြိုပျက်သွားသော RNA နမူနာပုံစံသည် cDNA ပေါင်းစပ်မှု၏ အထွက်နှုန်းနှင့် အရှည်ကို လျော့နည်းစေသည့် cDNA ပေါင်းစပ်မှုအတွက် ထိရောက်သော အလွှာတစ်ခုအဖြစ် အသုံးမပြုနိုင်တော့ပါ။ထို့ကြောင့်၊ reverse transcriptase ၏ RNaseH လုပ်ဆောင်ချက်ကို ဖယ်ရှားရန် သို့မဟုတ် လျှော့ချခြင်းသည် အကျိုးရှိမည်ဖြစ်သည်။
SuperScript Ⅱ reverse transcriptase၊ RNaseH- MMLV reverse transcriptase နှင့် thermoScript reverse transcriptase၊ RNaseH- AMV သည် MMLV နှင့် AMV ထက် ပိုမိုသောပမာဏနှင့် cDNA အပြည့်အစုံကို ရယူနိုင်ပါသည်။RT-PCR အာရုံခံနိုင်စွမ်းသည် cDNA ပေါင်းစပ်မှုပမာဏကြောင့် ထိခိုက်မည်ဖြစ်သည်။ThermoScript သည် AMV ထက် များစွာပို၍ ထိခိုက်လွယ်သည်။RT-PCR ထုတ်ကုန်များ၏ အရွယ်အစားသည် အထူးသဖြင့် ပိုမိုကြီးမားသော cDNA များကို ပုံတူပွားသည့်အခါတွင် cDNA ကို ပေါင်းစပ်ရန် ပြောင်းပြန် transcriptase စွမ်းရည်ဖြင့် ကန့်သတ်ထားသည်။MMLV နှင့် နှိုင်းယှဉ်ပါက SuperScripⅡသည် ရှည်လျားသော RT-PCR ထုတ်ကုန်များ၏ အထွက်နှုန်းကို သိသိသာသာ တိုးမြင့်စေသည်။RNaseH-reverse transcriptase သည် အပူချိန်ထိန်းနိုင်စွမ်း တိုးမြင့်လာသောကြောင့် တုံ့ပြန်မှုကို ပုံမှန် 37-42°C ထက် မြင့်မားသော အပူချိန်တွင် လုပ်ဆောင်နိုင်သည်။အကြံပြုထားသောပေါင်းစပ်မှုအခြေအနေများအောက်တွင်၊ oligo(dT) primer နှင့် [α-P]dCTP ၏ 10 μCi ကိုသုံးပါ။TCA မိုးရွာသွန်းမှုနည်းလမ်းဖြင့် ပထမကြိုး၏ စုစုပေါင်းအထွက်နှုန်းကို တွက်ချက်ခဲ့သည်။cDNA အရှည်ကို အရွယ်အစား-စီထားသော တီးဝိုင်းများကို ခွဲခြမ်းစိပ်ဖြာပြီး အယ်ကာလိုင်း agarose gel ပေါ်တွင် ရေတွက်ပြီး ခွဲခြမ်းစိတ်ဖြာထားပါသည်။
3. ပြောင်းပြန်ကူးယူမှုအတွက် ပေါက်ဖွားသည့်အပူချိန်ကို မြှင့်တင်ပါ-
ပိုမိုမြင့်မားသောပေါက်ဖွားမှုအပူချိန်သည် RNA အလယ်တန်းဖွဲ့စည်းပုံကိုဖွင့်ရန်ကူညီပေးသည်၊ တုံ့ပြန်မှု၏အထွက်နှုန်းကိုတိုးစေသည်။RNA နမူနာပုံစံအများစုအတွက် RNA နှင့် primers များကို ကြားခံ သို့မဟုတ် ဆားမပါဘဲ 65 ဒီဂရီစင်တီဂရိတ်တွင် ပေါက်ဖွားပြီး နောက်တွင် ရေခဲပေါ်တွင် လျှင်မြန်စွာအအေးခံခြင်းဖြင့် ဒုတိယဖွဲ့စည်းပုံအများစုကို ဖယ်ရှားပြီး primers များကို စည်းနိုင်စေမည်ဖြစ်သည်။သို့သော်၊ အချို့သောပုံစံများသည် အပူလွန်ကဲပြီးနောက်တွင်ပင် ဒုတိယတည်ဆောက်ပုံများရှိနေသေးသည်။ဤခက်ခဲသောပုံစံများကို ချဲ့ထွင်ခြင်းသည် ThermoScript Reverse Transcriptase ကို အသုံးပြု၍ ချဲ့ထွင်ခြင်းအား ပိုမိုကောင်းမွန်စေရန် မြင့်မားသောအပူချိန်တွင် ပြောင်းပြန်ကူးယူတုံ့ပြန်မှုကို ထားရှိနိုင်ပါသည်။အထူးသဖြင့် cDNA ပေါင်းစပ်မှုအတွက် gene-specific primers (GSP) ကိုအသုံးပြုသောအခါတွင် ပိုမိုမြင့်မားသော ပေါက်ဖွားမှုအပူချိန်များသည် တိကျမှုကို တိုးမြင့်စေနိုင်သည်။GSP ကိုအသုံးပြုပါက၊ primers များ၏ Tm သည် မျှော်လင့်ထားသည့် incubation temperature နှင့် တူညီကြောင်း သေချာပါစေ။60°C အထက် oligo(dT) နှင့် ကျပန်း primer များကို အသုံးမပြုပါနှင့်။ကျပန်း primers များသည် 60°C အထိမတိုးမီ 10 မိနစ်ကြာ 25°C တွင်ပေါက်ဖွားရန်လိုအပ်သည်။ပိုမိုမြင့်မားသော ပြောင်းပြန်ကူးယူဖော်ပြသည့်အပူချိန်ကိုအသုံးပြုခြင်းအပြင်၊ RNA/primer ရောနှောမှုကို 65°C denaturation temperature မှ reverse transcription incubation temperature သို့ တိုက်ရိုက်လွှဲပြောင်းပြီး pre-warmed 2 × တုံ့ပြန်မှုအရောအနှော (cDNA hot-start synthesis) ကို တိုက်ရိုက်လွှဲပြောင်းခြင်းဖြင့် တိကျမှုကို မြှင့်တင်နိုင်ပါသည်။ဤချဉ်းကပ်နည်းသည် အပူချိန်နိမ့်သည့်အချိန်တွင် ဖြစ်ပေါ်သော intermolecular base pairing ကို တားဆီးပေးသည်။RT-PCR အတွက် လိုအပ်သော အပူချိန်မျိုးစုံပြောင်းခြင်းကို အပူစက်ဘီးကို အသုံးပြုခြင်းဖြင့် ရိုးရှင်းနိုင်ပါသည်။
Tth thermostable polymerase သည် Mg2+ ၏ရှေ့မှောက်တွင် DNA polymerase အဖြစ်နှင့် Mn2+ တွင် RNA polymerase အဖြစ်လုပ်ဆောင်သည်။အမြင့်ဆုံးအပူချိန် 65 ဒီဂရီစင်တီဂရိတ်တွင် နွေးနိုင်ပါသည်။သို့ရာတွင်၊ PCR အတွင်း Mn2+ ပါဝင်မှုသည် သစ္စာရှိမှုကို လျော့နည်းစေပြီး၊ ၎င်းသည် cDNA ကိုပွားခြင်းကဲ့သို့သော တိကျသောအသံချဲ့ထွင်ခြင်းအတွက် Tth ပေါ်လီမာရတ်ကို လျော့နည်းစေသည်။ထို့အပြင်၊ Tth တွင် အာရုံခံနိုင်စွမ်းကို လျော့နည်းစေသည့် ပြောင်းပြန်ကူးယူဖော်ပြမှု ထိရောက်မှု နည်းပါးပြီး၊ ပြောင်းပြန်ကူးယူခြင်းနှင့် PCR အား အင်ဇိုင်းတစ်ခုတည်းဖြင့် လုပ်ဆောင်နိုင်သောကြောင့်၊ ပြောင်းပြန်ကူးယူခြင်းမရှိဘဲ ထိန်းချုပ်တုံ့ပြန်မှုများကို cDNA ချဲ့ထွင်ခြင်းထုတ်ကုန်များကို ညစ်ညမ်းစေသော DNA နှင့် နှိုင်းယှဉ်ရန် အသုံးမပြုနိုင်ပါ။အသံချဲ့စက် ထုတ်ကုန်များကို ခွဲခြားထားသည်။
4. ပြောင်းပြန်ကူးယူခြင်းကို မြှင့်တင်ပေးသည့် ပေါင်းထည့်မှုများ-
glycerol နှင့် DMSO အပါအဝင် additives များကို first-strand synthesis reaction တွင် ပေါင်းထည့်ထားပြီး၊ ၎င်းသည် nucleic acid နှစ်ထပ်ကြိုး၏ တည်ငြိမ်မှုကို လျှော့ချပေးပြီး RNA ၏ ဒုတိယဖွဲ့စည်းပုံကို ပြေလျော့စေနိုင်သည်။SuperScript II သို့မဟုတ် MMLV လုပ်ဆောင်ချက်ကို မထိခိုက်စေဘဲ 20% glycerol သို့မဟုတ် 10% DMSO အထိ ထည့်နိုင်သည်။AMV သည် လှုပ်ရှားမှုကို ဆုံးရှုံးခြင်းမရှိဘဲ 20% glycerol အထိ သည်းခံနိုင်သည်။SuperScriptⅡ reverse transcription တုံ့ပြန်မှုတွင် RT-PCR ၏ အာရုံခံနိုင်စွမ်းကို တိုးမြှင့်ရန်အတွက် 10% glycerol ကို 45°C တွင် ထည့်သွင်းပြီး ပျိုးထောင်နိုင်ပါသည်။ပြောင်းပြန်ကူးယူတုံ့ပြန်မှုထုတ်ကုန်၏ 1/10 ကို PCR သို့ပေါင်းထည့်ပါက၊ ချဲ့ထွင်မှုတုံ့ပြန်မှုတွင် glycerol ၏အာရုံစူးစိုက်မှုသည် PCR ကိုတားဆီးရန်မလုံလောက်ပါ။
5. RNaseH ကုသမှု
PCR မတိုင်မီ RNaseH နှင့် cDNA ပေါင်းစပ်တုံ့ပြန်မှုများကို ကုသခြင်းသည် အာရုံခံနိုင်စွမ်းကို တိုးစေနိုင်သည်။အချို့သောပုံစံများအတွက်၊ cDNA ပေါင်းစပ်မှုတုံ့ပြန်မှုတွင် RNA သည် အသံချဲ့ထွင်ခြင်းထုတ်ကုန်များ၏ ပေါင်းစပ်မှုကို တားဆီးပေးသည်၊ ယင်းအခြေအနေတွင် RNaseH ကုသမှုသည် အာရုံခံနိုင်စွမ်းကို တိုးမြင့်စေနိုင်သည်ဟု ယူဆပါသည်။ယေဘုယျအားဖြင့်၊ low-copy tuberous scherosis II ကဲ့သို့သော ပိုရှည်သော cDNA ပစ်မှတ်ပုံစံများကို ချဲ့ထွင်သည့်အခါ RNaseH ကုသမှုသည် လိုအပ်ပါသည်။ဤခက်ခဲသောပုံစံပုံစံအတွက်၊ RNaseH ကုသမှုသည် SuperScript II သို့မဟုတ် AMV-synthesized cDNA မှထုတ်လုပ်သည့်အချက်ပြမှုကို ပိုမိုကောင်းမွန်စေသည်။RT-PCR တုံ့ပြန်မှုအများစုအတွက်၊ RNaseH ကုသမှုသည် ရွေးချယ်နိုင်သည်၊ အကြောင်းမှာ PCR ၏အသွင်အပြင်အဆင့်သည် 95°C တွင် ယေဘုယျအားဖြင့် RNA:DNA complex ရှိ RNA ကို hydrolyzes ပေးသောကြောင့်ဖြစ်သည်။
6. အသေးစား RNA ထောက်လှမ်းခြင်းနည်းလမ်း
RNA အနည်းငယ်သာရရှိနိုင်သောအခါ RT-PCR သည် အထူးသဖြင့် စိန်ခေါ်မှုဖြစ်သည်။RNA သီးခြားခွဲထားစဉ်အတွင်း glycogen သည် သယ်ဆောင်သူအဖြစ် ပေါင်းထည့်ခြင်းဖြင့် နမူနာငယ်များ၏ အထွက်နှုန်းကို တိုးမြင့်စေပါသည်။RNase-free glycogen ကို Trizol ပေါင်းထည့်သကဲ့သို့ တစ်ချိန်တည်းတွင် ထည့်နိုင်သည်။Glycogen သည် ရေတွင်ပျော်ဝင်နိုင်ပြီး နောက်ဆက်တွဲမိုးရွာသွန်းမှုကို အထောက်အကူဖြစ်စေရန်အတွက် RNA ဖြင့် ရေတွင်ထိန်းသိမ်းထားနိုင်သည်။တစ်ရှူး ၅၀ မီလီဂရမ် သို့မဟုတ် မွေးမြူထားသောဆဲလ် ၁၀၆ ထက်နည်းသောနမူနာများအတွက်၊ အကြံပြုထားသည့် RNase-free glycogen ၏အာရုံစူးစိုက်မှုသည် 250 μg/ml ဖြစ်သည်။
SuperScript II ကို အသုံးပြု၍ ပြောင်းပြန် စာသားပြောင်းပြန်တုံ့ပြန်မှုတွင် acetylated BSA ပေါင်းထည့်ခြင်းသည် အာရုံခံနိုင်စွမ်းကို တိုးစေပြီး RNA ပမာဏအနည်းငယ်အတွက် SuperScript II ပမာဏကို လျှော့ချကာ RNaseOut nuclease inhibitor ယူနစ် 40 ပေါင်းထည့်ခြင်းဖြင့် ထောက်လှမ်းမှုအဆင့်ကို တိုးမြှင့်နိုင်သည်။RNA သီးခြားခွဲထုတ်ခြင်းလုပ်ငန်းစဉ်တွင် glycogen ကိုအသုံးပြုပါက၊ SuperScript II ကိုသုံးသောအခါတွင် BSA သို့မဟုတ် RNase inhibitor ကို ထပ်ထည့်ရန် အကြံပြုထားပါသည်။
二၊ RT-PCR တိကျမှုကို တိုးမြှင့်ပါ။
1. CND asynthesis-
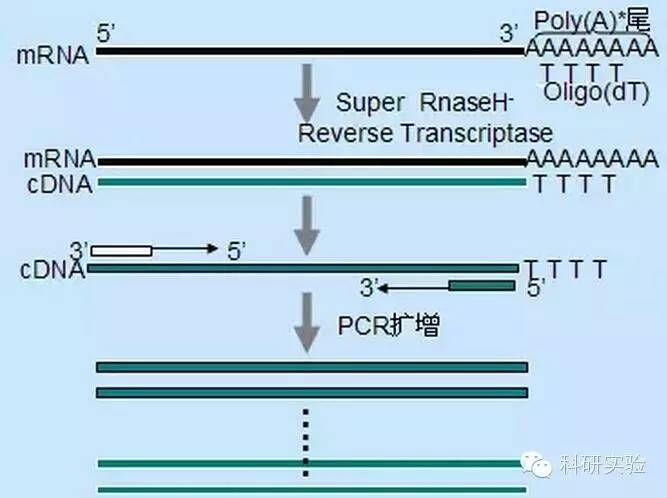
ပထမကြိုး cDNA ပေါင်းစပ်မှုကို မတူညီသော နည်းလမ်းသုံးမျိုးဖြင့် စတင်နိုင်သည်၊ ယင်း၏ ဆွေမျိုးတိကျမှုမှာ cDNA ပေါင်းစပ်ထားသော ပမာဏနှင့် အမျိုးအစားကို သက်ရောက်မှုရှိသည်။
ကျပန်း primer method သည် နည်းလမ်းသုံးမျိုးအနက် အတိအကျအနည်းဆုံးဖြစ်သည်။Primers များသည် စာသားမှတ်တမ်းတစ်လျှောက်ရှိ နေရာအများအပြားတွင် တိုတောင်းသော၊ တစ်စိတ်တစ်ပိုင်း-အရှည် cDNA များကိုထုတ်ပေးသည်။ဤနည်းလမ်းကို 5′ အဆုံးအစီအစဥ်များရယူရန်နှင့် အလယ်တန်းဖွဲ့စည်းပုံ၏ဒေသများနှင့်အတူ RNA နမူနာများမှ cDNA ရယူရန် သို့မဟုတ် reverse transcriptase ဖြင့် ပုံတူပွားမရနိုင်သော termination sites များနှင့် မကြာခဏအသုံးပြုလေ့ရှိသည်။အရှည်ဆုံး cDNA ကိုရရှိရန်၊ RNA နမူနာတစ်ခုစီရှိ primers နှင့် RNA အချိုးကို မျက်မြင်ကိုယ်တွေ့ ဆုံးဖြတ်ရန် လိုအပ်သည်။ကျပန်း primers များ၏စတင်အာရုံစူးစိုက်မှုသည် 20 μlတုံ့ပြန်မှုတစ်ခုလျှင် 50 မှ 250 ng မှအကွာအဝေးရှိသည်။ကျပန်း primers များကို အသုံးပြု၍ cDNA သည် စုစုပေါင်း RNA မှ ပေါင်းစပ်ထုတ်လုပ်ထားသော ribosomal RNA ဖြစ်သောကြောင့်၊ poly(A)+RNA ကို ယေဘုယျအားဖြင့် ပုံစံပလိတ်အဖြစ် ရွေးချယ်သည်။
Oligo(dT) primers များသည် ကျပန်း primers များထက် ပိုမိုတိကျပါသည်။၎င်းသည် eukaryotic mRNAs အများစု၏ 3′ အဆုံးတွင်တွေ့ရသော poly(A) အမြီးသို့ ပေါင်းစပ်သွားပါသည်။poly(A)+ RNA သည် စုစုပေါင်း RNA ၏ ခန့်မှန်းခြေ 1% မှ 2% ဖြစ်သောကြောင့်၊ cDNA ၏ ပမာဏနှင့် ရှုပ်ထွေးမှုသည် ကျပန်း primers များထက် များစွာနည်းပါသည်။၎င်း၏မြင့်မားသောတိကျမှုများကြောင့်၊ oligo(dT) သည် ယေဘုယျအားဖြင့် RNA နှင့် primers နှင့် poly(A)+ ရွေးချယ်မှုအချိုးကို ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ရန်မလိုအပ်ပါ။20μl တုံ့ပြန်မှုစနစ်အတွက် 0.5μg oligo(dT) ကို အသုံးပြုရန် အကြံပြုထားသည်။oligo(dT)12-18 သည် RT-PCR အများစုအတွက် သင့်လျော်သည်။ThermoScript RT-PCR စနစ်သည် ပိုမိုမြင့်မားသောပေါက်ဖွားမှုအပူချိန်အတွက် ၎င်း၏ပိုမိုကောင်းမွန်သောအပူတည်ငြိမ်မှုကြောင့် oligo(dT)20 ကို ပေးဆောင်သည်။
Gene specific primers (GSP) များသည် reverse transcription step အတွက် အတိကျဆုံး primers များဖြစ်သည်။GSP သည် RNA အားလုံးကို ပေါင်းစည်းထားသည့် ကျပန်း primers သို့မဟုတ် oligo(dT) နှင့် မတူဘဲ အထူးသဖြင့် RNA ပစ်မှတ်အစီအစဉ်သို့ ပေါင်းစပ်နိုင်သော antisense oligonucleotide တစ်ခုဖြစ်သည်။PCR primer များကိုဒီဇိုင်းဆွဲရာတွင်အသုံးပြုသည့်တူညီသောစည်းမျဉ်းများသည် reverse transcription reactions တွင် GSP ၏ဒီဇိုင်းနှင့်သက်ဆိုင်ပါသည်။GSP သည် mRNA ၏ 3′-mRNA ၏အစွန်းဆုံးသို့ ချိတ်ဆက်သည့် အသံချဲ့စက်နှင့် တူညီသော sequence ဖြစ်နိုင်သည်၊ သို့မဟုတ် GSP သည် reverse amplification primer ၏ အောက်ပိုင်းကို ထည့်သွင်းရန် ဒီဇိုင်းထုတ်နိုင်သည်။အချို့သော ချဲ့ထွင်ထားသော အကြောင်းအရာများအတွက်၊ ပစ်မှတ် RNA ၏ ဒုတိယဖွဲ့စည်းပုံသည် primer binding ကို တားဆီးနိုင်သောကြောင့် အောင်မြင်သော RT-PCR အတွက် antisense primer တစ်ခုထက်ပိုသော ဒီဇိုင်းပြုလုပ်ရန် လိုအပ်ပါသည်။20 μl ပထမကြိုးပေါင်းစပ်မှုတုံ့ပြန်မှုတွင် 1 pmol antisense GSP ကိုအသုံးပြုရန် အကြံပြုထားသည်။
2. ပြောင်းပြန်ကူးယူမှုအတွက် ပေါက်ဖွားသည့်အပူချိန်ကို မြှင့်တင်ပါ-
GSP သတ်မှတ်မှု၏ အပြည့်အဝအားသာချက်ကို အပြည့်အဝအသုံးချနိုင်ရန်၊ ပိုမိုမြင့်မားသော အပူချိန်ထိန်းနိုင်သော ပြောင်းပြန်စာသားမှတ်တမ်းကို အသုံးပြုသင့်သည်။တုံ့ပြန်မှုတင်းမာမှုကို တိုးမြှင့်ရန်အတွက် အပူခံနိုင်သော ပြောင်းပြန် စာသားမှတ်တမ်းများကို ပိုမိုမြင့်မားသော အပူချိန်တွင် မွေးမြူနိုင်သည်။ဥပမာအားဖြင့်၊ GSP တစ်ခုသည် 55°C တွင် တိုးလာပါက၊ AMV သို့မဟုတ် M-MLV အား တင်းကျပ်မှုနည်းသော 37°C တွင် ပြောင်းပြန်ကူးယူခြင်းအတွက် အသုံးပြုပါက GSP ၏ တိကျသေချာမှုကို အပြည့်အဝအသုံးချမည်မဟုတ်ပါ။သို့သော်လည်း၊ SuperScript II နှင့် ThermoScript သည် 50°C နှင့်အထက်တွင် တုံ့ပြန်နိုင်ပြီး၊ ၎င်းသည် အပူချိန်နိမ့်သောနေရာတွင် ထုတ်ပေးသော သီးခြားမဟုတ်သော ထုတ်ကုန်များကို ဖယ်ရှားပစ်မည်ဖြစ်သည်။အမြင့်ဆုံးတိကျမှုအတွက်၊ RNA/primer ရောနှောခြင်းကို 65°C denaturation temperature မှ reverse transcription incubation temperature သို့ တိုက်ရိုက်လွှဲပြောင်းနိုင်ပြီး pre-warmed 2× reaction mix (cDNA synthesis hot start) သို့ ပေါင်းထည့်နိုင်သည်။၎င်းသည် အပူချိန်နိမ့်သောအချိန်တွင် intermolecular base တွဲခြင်းကို တားဆီးရန် ကူညီပေးသည်။RT-PCR အတွက် လိုအပ်သော အပူချိန်အကူးအပြောင်းများစွာကို အပူစက်ဘီးကို အသုံးပြုခြင်းဖြင့် ရိုးရှင်းစေနိုင်သည်။
3. မျိုးဗီဇ DNA ညစ်ညမ်းမှုကို လျှော့ချပေးသည်-
RT-PCR တွင် ကြုံတွေ့နိုင်သည့် အလားအလာ အခက်အခဲမှာ RNA ရှိ မျိုးရိုးဗီဇ DNA ညစ်ညမ်းခြင်း ဖြစ်သည်။Trizol Reagent ကဲ့သို့သော ကောင်းမွန်သော RNA သီးခြားခွဲထုတ်နည်းကို အသုံးပြုခြင်းဖြင့် RNA ပြင်ဆင်မှုကို ညစ်ညမ်းစေသော မျိုးရိုးဗီဇ DNA ပမာဏကို လျှော့ချနိုင်မည်ဖြစ်သည်။မျိုးရိုးဗီဇ DNA မှရရှိသော ထုတ်ကုန်များကို ရှောင်ရှားရန်၊ RNA သည် ညစ်ညမ်းနေသော DNA ကို ပြောင်းပြန်မကူးမီ ဖယ်ရှားရန် အသံချဲ့စက်-အဆင့် DNase I ဖြင့် ကုသနိုင်ပါသည်။2.0 mM EDTA တွင် နမူနာများကို 65°C တွင် 10 မိနစ်ကြာ ပျိုးထောင်ခြင်းဖြင့် DNase I အစာခြေခြင်းကို ရပ်တန့်ခဲ့သည်။EDTA သည် မြင့်မားသောအပူချိန်တွင် မဂ္ဂနီဆီယမ်အိုင်းယွန်းများကို chelate လုပ်နိုင်ပြီး magnesium ion-dependent RNA hydrolysis ကို တားဆီးပေးသည်။
ချဲ့ထွင်ထားသော cDNA ကို genomic DNA amplification ထုတ်ကုန်များ ညစ်ညမ်းစေခြင်းမှ ခွဲထုတ်ရန်အတွက်၊ anneal တစ်ခုစီသည် exons ကို ခွဲခြားရန် primers ကို ဒီဇိုင်းထုတ်နိုင်သည်။cDNA မှရရှိသော PCR ထုတ်ကုန်များသည် ညစ်ညမ်းနေသော မျိုးရိုးဗီဇ DNA မှရရှိသော ထုတ်ကုန်များထက် ပိုတိုလိမ့်မည်။ထို့အပြင်၊ ပေးထားသောအပိုင်းအစသည် မျိုးရိုးဗီဇ DNA သို့မဟုတ် cDNA မှဆင်းသက်လာခြင်းရှိမရှိ ဆုံးဖြတ်ရန် RNA ပုံစံတစ်ခုစီတွင် ပြောင်းပြန်ကူးယူခြင်းမပြုဘဲ ထိန်းချုပ်စမ်းသပ်မှုတစ်ခုကို ပြုလုပ်ခဲ့သည်။ပြောင်းပြန် ကူးယူဖော်ပြခြင်းမရှိဘဲ ရရှိသော PCR ထုတ်ကုန်သည် ဂျီနိုမ်မှ ဆင်းသက်လာသည်။
စာတိုက်အချိန်- မေ ၁၆-၂၀၂၃








