- PCR သည် DNA နမူနာပုံစံအနည်းငယ်မှ DNA ကိုချဲ့ထွင်ရန်အသုံးပြုသည့်နည်းလမ်းတစ်ခုဖြစ်သည်။RT-PCR သည် ချဲ့ထွင်နိုင်သည့် RNA ရင်းမြစ်မှ DNA ပုံစံတစ်ခုကို ထုတ်လုပ်ရန် ပြောင်းပြန် စာသားကို အသုံးပြုသည်။
- PCR နှင့် RT-PCR တို့သည် ပုံမှန်အားဖြင့် အဆုံးမှတ်တုံ့ပြန်မှုများဖြစ်ပြီး qPCR နှင့် RT-qPCR တို့သည် PCR တုံ့ပြန်မှုအတွင်း ထုတ်ကုန်ပေါင်းစပ်မှုနှုန်း၏ kinetics ကို အသုံးပြု၍ ပုံစံပလိတ်ပမာဏကို quantitate ပြုလုပ်ရန် အသုံးပြုသည်။
- ဒစ်ဂျစ်တယ် PCR ကဲ့သို့သော အသစ်သောနည်းလမ်းများသည် ကနဦး DNA နမူနာပုံစံ၏ ပကတိအရေအတွက်ကို ပေးစွမ်းနိုင်သော်လည်း isothermal PCR ကဲ့သို့သော နည်းလမ်းများသည် ယုံကြည်စိတ်ချရသော ရလဒ်များကို ပေးဆောင်ရန် ဈေးကြီးသော စက်ကိရိယာများ လိုအပ်မှုကို လျော့နည်းစေသည်။
Polymerase ကွင်းဆက်တုံ့ပြန်မှု (PCR) သည် DNA နှင့် RNA sequence များကို ချဲ့ထွင်သိရှိနိုင်စေရန် အတော်လေးရိုးရှင်းပြီး အသုံးများသော မော်လီကျူးဇီဝဗေဒနည်းပညာတစ်ခုဖြစ်သည်။ရက်ပေါင်းများစွာ ကြာတတ်သည့် DNA ပွားခြင်းနှင့် ချဲ့ထွင်ခြင်း၏ ရိုးရာနည်းလမ်းများနှင့် နှိုင်းယှဉ်ပါက PCR သည် နာရီအနည်းငယ်သာ လိုအပ်ပါသည်။PCR သည် အလွန်အကဲဆတ်ပြီး သီးခြား sequence များကို ရှာဖွေတွေ့ရှိရန်နှင့် ချဲ့ထွင်ရန်အတွက် အနည်းငယ်မျှသာ နမူနာပုံစံ လိုအပ်ပါသည်။အခြေခံ PCR နည်းလမ်းများသည် ရိုးရှင်းသော DNA နှင့် RNA ထောက်လှမ်းခြင်းမှ ပိုမိုအဆင့်မြင့်လာသည်။အောက်တွင်၊ ကျွန်ုပ်တို့သည် သင့်သုတေသနလိုအပ်ချက်အတွက် Enzo Life Sciences တွင် ကျွန်ုပ်တို့ပေးဆောင်သော မတူညီသော PCR နည်းလမ်းများနှင့် ဓာတုပစ္စည်းများ၏ ခြုံငုံသုံးသပ်ချက်ကို ပေးထားပါသည်။ကျွန်ုပ်တို့သည် သိပ္ပံပညာရှင်များအား ၎င်းတို့၏နောက်ထပ်သုတေသနပရောဂျက်တွင် အသုံးပြုရန် PCR ဓာတ်ပစ္စည်းများကို လျင်မြန်စွာဝင်ရောက်ကူညီရန် ရည်ရွယ်ပါသည်။
PCR
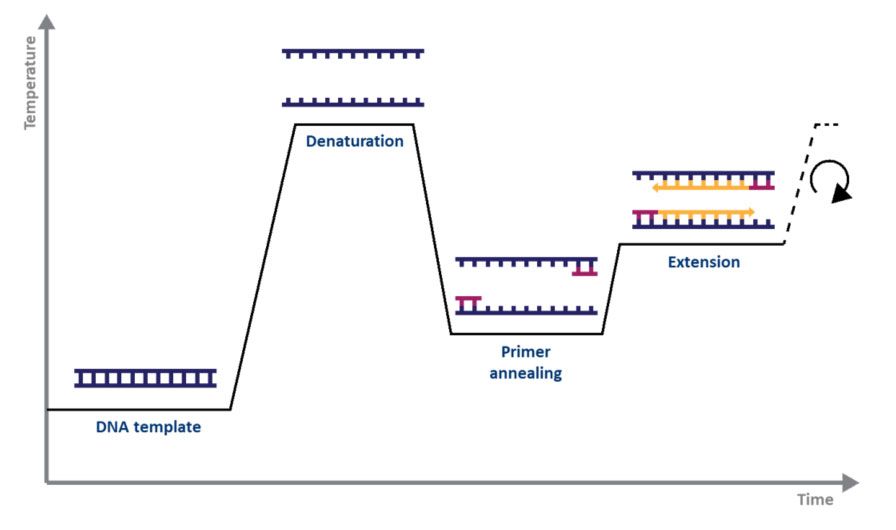
ပုံမှန် PCR အတွက်၊ သင်လိုအပ်သမျှမှာ DNA polymerase၊ မဂ္ဂနီဆီယမ်၊ nucleotides၊ primers၊ ချဲ့ထွင်ရန် DNA ပုံစံခွက်၊ နှင့် thermocycler တစ်ခုဖြစ်သည်။PCR ယန္တရားသည် ၎င်း၏ရည်ရွယ်ချက်အတိုင်း ရိုးရှင်းသည်- 1) double-stranded DNA (dsDNA) သည် အပူရှိန်မရှိ၊ 2) primers များသည် single DNA strands နှင့် လိုက်လျောညီထွေဖြစ်ပြီး 3) primer များကို DNA polymerase ဖြင့် တိုးချဲ့ထားသောကြောင့် မိတ္တူနှစ်စောင်ကို ထုတ်ပေးပါသည်။ မူရင်း DNA ကြိုး။အပူချိန်နှင့် အကြိမ်များစွာတွင် denaturation, annealing, နှင့် elongation process ကို ချဲ့ထွင်ခြင်း စက်ဝန်းတစ်ခု (ပုံ. 1) ဟုခေါ်သည်။
| ပုံ 1။PCR မှ ချဲ့ထွင်ခြင်း စက်ဝန်း၏ ဇယားကွက်ကို ကိုယ်စားပြုခြင်း။ |
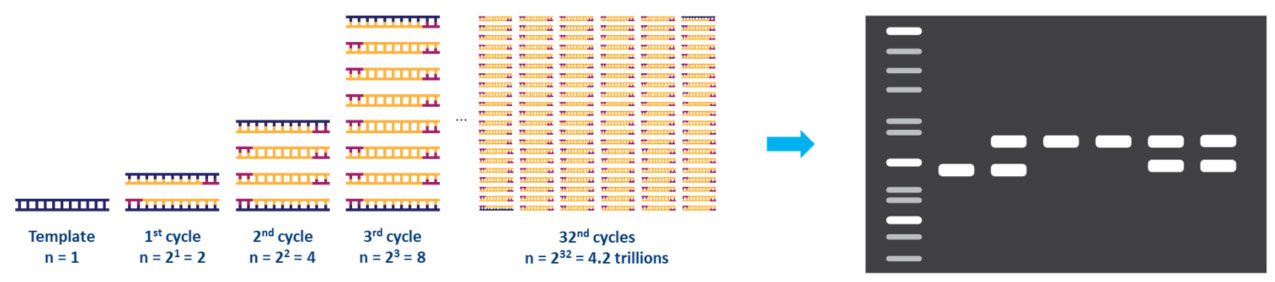
စက်ဝန်း၏ အဆင့်တစ်ဆင့်ချင်းစီကို အသုံးပြုထားသော ပုံစံခွက်နှင့် primer အစုံအတွက် အကောင်းဆုံးဖြစ်အောင် ပြုလုပ်သင့်သည်။ဤစက်ဝန်းကို အကြိမ် 20-40 ခန့် ထပ်ခါတလဲလဲ ပြုလုပ်ပြီး ချဲ့ထွင်ထားသော ထုတ်ကုန်ကို ပုံမှန်အားဖြင့် agarose gel (ပုံ 2) ဖြင့် ခွဲခြမ်းစိတ်ဖြာနိုင်ပါသည်။
| ပုံ ၂။PCR ဖြင့် DNA နမူနာပုံစံကို ချဲ့ထွင်ခြင်းနှင့် agarose gel electrophoresis ဖြင့် ခွဲခြမ်းစိတ်ဖြာခြင်း။ |
PCR သည် အလွန်အထိခိုက်မခံသောနည်းလမ်းဖြစ်ပြီး တုံ့ပြန်မှုတစ်ခုတည်းအတွက် အလွန်သေးငယ်သောပမာဏလိုအပ်သောကြောင့်၊ တုံ့ပြန်မှုများစွာအတွက် မာစတာရောနှောပြင်ဆင်မှုကို အကြံပြုထားသည်။မာစတာရောနှောခြင်းကို ကောင်းစွာရောစပ်ပြီး တုံ့ပြန်မှုအရေအတွက်အလိုက် ပိုင်းခြားရမည်ဖြစ်ပြီး တုံ့ပြန်မှုတစ်ခုစီတွင် တူညီသောအင်ဇိုင်း၊ dNTPs နှင့် primers ပမာဏများပါ၀င်ကြောင်း သေချာစေမည်ဖြစ်သည်။Enzo Life Sciences ကဲ့သို့သော ပေးသွင်းသူများစွာသည် primers နှင့် DNA template မှလွဲ၍ အရာအားလုံးပါဝင်ပြီးသားဖြစ်သော PCR ရောနှောမှုကိုလည်း ပေးဆောင်ပါသည်။
Guanine/Cytosine-rich (GC-rich) ဒေသများသည် စံ PCR နည်းပညာများတွင် စိန်ခေါ်မှုတစ်ရပ်ကို ကိုယ်စားပြုသည်။GC ကြွယ်ဝသော အတွဲများသည် GC ပါဝင်မှုနည်းသော အတွဲများထက် ပိုမိုတည်ငြိမ်သည်။ထို့အပြင်၊ GC ကြွယ်ဝသော sequences များသည် hairpin loops ကဲ့သို့သော ဒုတိယတည်ဆောက်ပုံများကို ဖြစ်ပေါ်စေပါသည်။ရလဒ်အနေဖြင့်၊ GC ကြွယ်ဝသောနှစ်ထပ်ကြိုးများသည် denaturation အဆင့်အတွင်း လုံးဝခွဲရန်ခက်ခဲသည်။ထို့ကြောင့် DNA polymerase သည် အတားအဆီးမရှိဘဲ ကြိုးအသစ်ကို ပေါင်းစပ်၍မရနိုင်ပါ။ပိုမိုမြင့်မားသော denaturation အပူချိန်သည် ၎င်းကို ပိုမိုကောင်းမွန်လာစေနိုင်ပြီး ပိုမိုမြင့်မားသော annealing အပူချိန်နှင့် တိုတောင်းသော annealing time သို့ ချိန်ညှိမှုများသည် GC ကြွယ်ဝသော primers များ၏ တိကျသောစည်းနှောင်မှုကို တားဆီးနိုင်သည်။အပိုဓာတ်ပစ္စည်းများသည် GC ကြွယ်ဝသော sequences များ၏ ချဲ့ထွင်မှုကို မြှင့်တင်နိုင်သည်။DMSO၊ glycerol နှင့် betaine တို့သည် GC အပြန်အလှန်ဆက်သွယ်မှုများကြောင့် ဖြစ်ပေါ်လာသော ဒုတိယဖွဲ့စည်းပုံများကို နှောင့်ယှက်ရန် ကူညီပေးပြီး နှစ်ဆမျှင်များကို ခွဲထုတ်ရာတွင် လွယ်ကူချောမွေ့စေပါသည်။
Hot Start PCR
တိကျသော အသံချဲ့စက်သည် PCR အတွင်း ဖြစ်ပွားနိုင်သည့် ပြဿနာတစ်ခုဖြစ်သည်။PCR အတွက်အသုံးပြုသော DNA ပေါ်လီမာအများစုသည် အပူချိန် 68°C မှ 72°C အတွင်းတွင် အကောင်းဆုံးအလုပ်လုပ်သည်။အင်ဇိုင်းသည် အပူချိန်နိမ့်သော်လည်း အပူချိန်နိမ့်သည့်နေရာတွင်လည်း လုပ်ဆောင်နိုင်သည်။လိမ်းထားသော အပူချိန်အောက် အလှမ်းဝေးသော အပူချိန်တွင်၊ primers များသည် အတိအကျမဟုတ်သော ချည်နှောင်နိုင်ပြီး ရေခဲပေါ်တွင် တုံ့ပြန်မှုပြုလုပ်ထားလျှင်ပင် အတိအကျမဟုတ်သော ချဲ့ထွင်မှုဆီသို့ ဦးတည်သွားနိုင်သည်။အချို့သောအပူချိန်ရောက်သည်နှင့်တစ်ပြိုင်နက် DNA polymerase မှခွဲထွက်သော polymerase inhibitors များကိုအသုံးပြုခြင်းဖြင့်၎င်းကိုကာကွယ်နိုင်သည်၊ ထို့ကြောင့် hot start PCR ဟူသောအသုံးအနှုန်းကိုအသုံးပြုခြင်းဖြင့်ကာကွယ်နိုင်သည်။inhibitor သည် ကနဦး denaturation temperature (ပုံမှန်အားဖြင့် 95°C) တွင် polymerase နှင့် denatures များကို ချည်နှောင်ထားသည့် ပဋိပစ္စည်းဖြစ်နိုင်သည်။
မြင့်မားသော Fidelity Polymerase
DNA ပေါ်လီမာရတ်များသည် မူရင်းပုံစံပလိတ်အစီအစဥ်ကို တိကျစွာ ချဲ့ထွင်ထားသော်လည်း နျူကလီးအိုတိုက်နှင့် ကိုက်ညီမှုတွင် အမှားအယွင်းများ ဖြစ်ပေါ်လာနိုင်သည်။ပုံတူပွားခြင်းကဲ့သို့သော အပလီကေးရှင်းများတွင် မကိုက်ညီမှုများသည် ဖြတ်တောက်ထားသော စာသားများကို ဖြစ်ပေါ်စေနိုင်ပြီး ဘာသာပြန်မှားသော သို့မဟုတ် မလှုပ်ရှားနိုင်သော ပရိုတိန်းများကို ရေအောက်ပိုင်းတွင် ဖြစ်စေနိုင်သည်။ဤမတူညီမှုများကို ရှောင်ရှားရန်၊ "စစ်ဖတ်ခြင်း" လုပ်ဆောင်ချက်ပါရှိသော ပိုလီမာရတ်များကို အလုပ်အသွားအလာတွင် ဖော်ထုတ်ပြီး ထည့်သွင်းထားသည်။ပထမဆုံး ပေါ်လီမာရတ် (Pfu) ကို ၁၉၉၁ ခုနှစ်တွင် Pyrococcus furiosus တွင် တွေ့ရှိခဲ့သည်။ဤ Pfu အင်ဇိုင်းသည် 3' မှ 5' exonuclease လုပ်ဆောင်မှုရှိသည်။DNA ကို ချဲ့ထွင်လိုက်သည်နှင့်အမျှ၊ exonuclease သည် ကြိုး၏ 3' စွန်းတွင် မကိုက်ညီသော nucleotides များကို ဖယ်ရှားသည်။မှန်ကန်သော nucleotide ကို အစားထိုးပြီး DNA ပေါင်းစပ်မှုကို ဆက်လက်လုပ်ဆောင်သည်။မှားယွင်းသော နျူကလီးအိုတိုက် စည်းရိုးများကို ဖော်ထုတ်ခြင်းသည် မှန်ကန်သော nucleoside triphosphate အင်ဇိုင်းနှင့် ဆက်စပ်မှုအပေါ် အခြေခံထားခြင်းဖြစ်ပြီး၊ ထိရောက်မှုမရှိသော ပေါင်းစပ်ပေါင်းစပ်မှုသည် ပေါင်းစပ်မှုကို နှေးကွေးစေပြီး မှန်ကန်သော အစားထိုးမှုကို ခွင့်ပြုပေးပါသည်။Pfu polymerase ၏ အထောက်အထားဖတ်ခြင်းလုပ်ဆောင်ချက်သည် Taq DNA polymerase နှင့် နှိုင်းယှဉ်ပါက နောက်ဆုံးအဆင့်တွင် အမှားအယွင်းနည်းသည်။မကြာသေးမီနှစ်များအတွင်း၊ အခြားသော အထောက်အထားဖတ်ခြင်းအင်ဇိုင်းများကို ဖော်ထုတ်ခဲ့ပြီး DNA ချဲ့ထွင်မှုအတွင်း အမှားအယွင်းနှုန်းကို ထပ်မံလျှော့ချရန်အတွက် မူလ Pfu အင်ဇိုင်းများကို ပြုပြင်မွမ်းမံမှုများ ပြုလုပ်ခဲ့သည်။
RT-PCR
Reverse transcription PCR သို့မဟုတ် RT-PCR သည် RNA ကို ပုံစံပလိတ်အဖြစ် အသုံးပြုခွင့်ပေးသည်။နောက်ထပ်အဆင့်တစ်ခုသည် RNA ၏ရှာဖွေတွေ့ရှိမှုနှင့်ချဲ့ထွင်မှုကိုခွင့်ပြုသည်။RNA သည် reverse transcriptase ကို အသုံးပြု၍ ဖြည့်စွက် DNA (cDNA) အဖြစ် ပြောင်းပြန် ကူးယူပါသည်။RNA နမူနာပုံစံ၏ အရည်အသွေးနှင့် သန့်ရှင်းမှုသည် RT-PCR အောင်မြင်မှုအတွက် မရှိမဖြစ်လိုအပ်သည်။RT-PCR ၏ပထမအဆင့်သည် DNA/RNA ပေါင်းစပ်ပေါင်းစပ်မှုဖြစ်သည်။Reverse transcriptase တွင် ဟိုက်ဘရစ်၏ RNA အပိုင်းကို ကျဆင်းစေသည့် RNase H လုပ်ဆောင်ချက်လည်း ပါရှိသည်။ထို့နောက် သောင်တင်ထားသော DNA မော်လီကျူးသည် DNA-မူတည်သည့် DNA polymerase လုပ်ဆောင်မှုဖြင့် cDNA သို့ ပြောင်းပြန် transcriptase လုပ်ဆောင်ချက်ဖြင့် ပြီးမြောက်သည်။ပထမကြိုးမျှင်တုံ့ပြန်မှု၏ ထိရောက်မှုသည် ချဲ့ထွင်ခြင်းလုပ်ငန်းစဉ်ကို ထိခိုက်စေနိုင်သည်။ဤနေရာမှစ၍၊ စံ PCR လုပ်ထုံးလုပ်နည်းကို cDNA ချဲ့ထွင်ရန် အသုံးပြုသည်။RT-PCR မှ RNA ကို cDNA သို့ပြန်ပြောင်းရန်ဖြစ်နိုင်ခြေသည် အားသာချက်များစွာရှိပြီး ၎င်းကို မျိုးရိုးဗီဇဖော်ပြမှုခွဲခြမ်းစိတ်ဖြာမှုတွင် အဓိကအသုံးပြုသည်။RNA သည် single-stranded ဖြစ်ပြီး အလွန်မတည်မငြိမ်ဖြစ်ပြီး ၎င်းသည် ၎င်းနှင့်အလုပ်လုပ်ရန် စိန်ခေါ်မှုဖြစ်စေသည်။၎င်းသည် ဇီဝဗေဒနမူနာတစ်ခုတွင် RNA မှတ်တမ်းများကို တိုင်းတာသည့် qPCR ၏ ပထမအဆင့်အဖြစ် လုပ်ဆောင်လေ့ရှိသည်။
qPCR နှင့် RT-qPCR
Quantitative PCR (qPCR) ကို အသုံးချမှု အများအပြားအတွက် nucleic acids များကို ရှာဖွေရန်၊ လက္ခဏာရပ်နှင့် အရေအတွက် တိုင်းတာရန်အတွက် အသုံးပြုပါသည်။RT-qPCR တွင်၊ RNA မှတ်တမ်းများကို အထက်တွင်ဖော်ပြထားသည့်အတိုင်း cDNA သို့ ပထမဦးစွာ ပြောင်းပြန်ကူးယူခြင်းဖြင့် မကြာခဏ အရေအတွက်ကို တိုင်းတာပြီး၊ ထို့နောက် qPCR ကို နောက်ပိုင်းတွင် လုပ်ဆောင်သည်။ပုံမှန် PCR တွင်ကဲ့သို့ပင်၊ DNA ကို ထပ်ခါတလဲလဲ အဆင့်သုံးဆင့်ဖြင့် ချဲ့ထွင်သည်- denaturation၊ annealing နှင့် elongation။သို့သော်၊ qPCR တွင်၊ အလင်းတန်းတံဆိပ်တပ်ခြင်းသည် PCR တိုးတက်လာသည်နှင့်အမျှ ဒေတာစုဆောင်းမှုကို လုပ်ဆောင်နိုင်သည်။ဤနည်းပညာသည် ရနိုင်သောနည်းလမ်းများနှင့် ဓာတုဗေဒနည်းများဖြင့် အကျိုးကျေးဇူးများစွာရှိသည်။
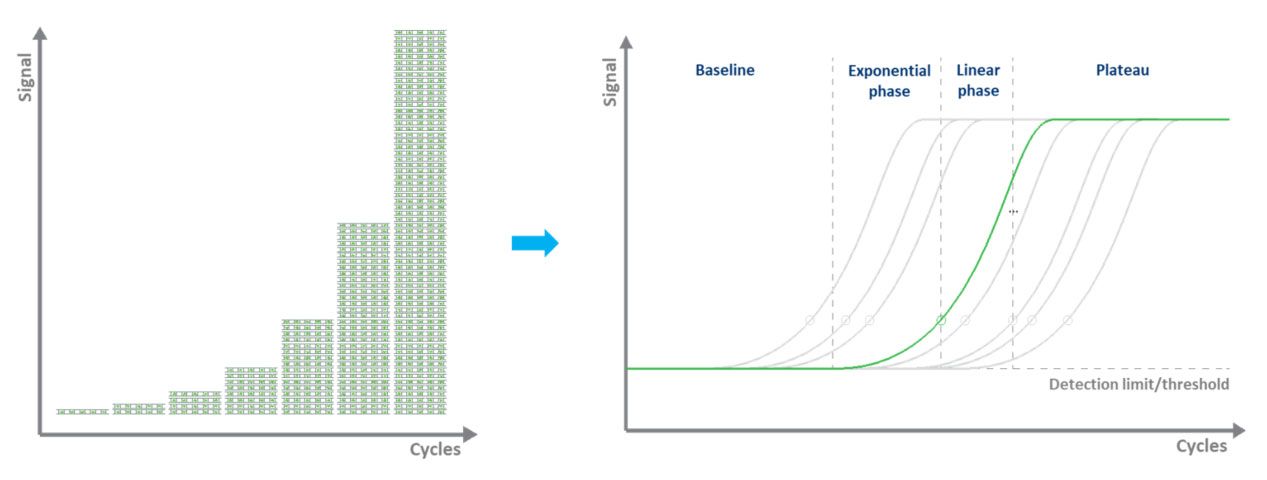
ဆိုးဆေးအခြေခံ qPCR (ပုံမှန်အားဖြင့် အစိမ်း) တွင်၊ မီးချောင်းတံဆိပ်ကပ်ခြင်းသည် dsDNA ပေါင်းစပ်ဆိုးဆေးကိုအသုံးပြုခြင်းဖြင့် ချဲ့ထွင်ထားသော DNA မော်လီကျူးများ၏ အရေအတွက်ကို အတိုင်းအတာတစ်ခုအထိ ခွင့်ပြုပေးသည်။စက်ဝန်းတစ်ခုစီတွင် fluorescence ကို တိုင်းတာသည်။fluorescence အချက်ပြမှုသည် ထပ်တူပြုထားသော DNA ပမာဏနှင့် အချိုးကျ တိုးလာသည်။ထို့ကြောင့်၊ DNA ကို “အချိန်နှင့်တပြေးညီ” ဖြင့် တိုင်းတာသည် (ပုံ ၃)။ဆိုးဆေးအခြေခံ qPCR ၏ အားနည်းချက်များမှာ တစ်ကြိမ်လျှင် ပစ်မှတ်တစ်ခုသာ စစ်ဆေးနိုင်ပြီး ဆိုးဆေးသည် နမူနာတွင်ပါရှိသော ds-DNA နှင့် ချည်နှောင်မည် ဖြစ်သည်။
| ပုံ ၃။qPCR ဖြင့် DNA နမူနာပုံစံကို ချဲ့ထွင်ပြီး fluorescence အချက်ပြမှုကို အချိန်နှင့်တပြေးညီ တိုင်းတာခြင်း။ |
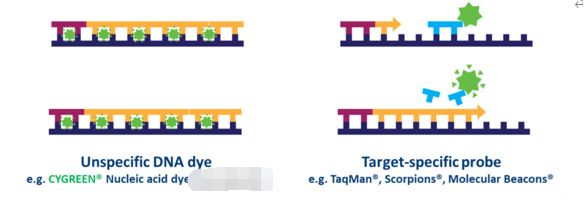
probe-based qPCR တွင်၊ နမူနာတစ်ခုစီတွင် ပစ်မှတ်အများအပြားကို တစ်ပြိုင်နက် ရှာဖွေတွေ့ရှိနိုင်သော်လည်း ၎င်းသည် primers များအပြင် အသုံးပြုသည့် ပစ်မှတ်အလိုက် တိကျသည့် probe(များ) ကို ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ရန်နှင့် ဒီဇိုင်းပြုလုပ်ရန် လိုအပ်ပါသည်။Probe ဒီဇိုင်း အမျိုးအစားများစွာကို ရနိုင်သော်လည်း အသုံးအများဆုံး အမျိုးအစားမှာ fluorophore နှင့် quencher ပေါင်းစပ်ထားသည့် hydrolysis probe ဖြစ်သည်။Fluorescence resonance energy transfer (FRET) သည် probe သည် နဂိုအတိုင်းရှိနေချိန်တွင် quencher မှတစ်ဆင့် fluorophore ၏ ထုတ်လွှတ်မှုကို တားဆီးပေးသည်။သို့ရာတွင်၊ PCR တုံ့ပြန်မှုအတွင်း၊ ပလေယာကို primer တိုးချဲ့မှုနှင့် ၎င်းနှင့်ချိတ်ဆက်ထားသော သီးခြား sequence ၏ ချဲ့ထွင်မှုအတွင်း စူးစမ်းလေ့လာမှုကို ဟိုက်ဒရောလစ်ဖြစ်စေသည်။probe ၏ ကွဲထွက်မှုသည် fluorophore ကို quencher နှင့် ပိုင်းခြားပြီး ချဲ့ထွင်မှုအပေါ် မူတည်ပြီး fluorescence တိုးလာသည် (ပုံ. 4)။ထို့ကြောင့်၊ probe-based qPCR တုံ့ပြန်မှုမှ fluorescence signal သည် နမူနာတွင်ရှိသော probe target sequence ပမာဏနှင့် အချိုးကျပါသည်။probe-based qPCR သည် ဆိုးဆေးအခြေခံ qPCR ထက် ပိုမိုတိကျသောကြောင့်၊ ၎င်းသည် qPCR-based ရောဂါရှာဖွေစစ်ဆေးမှုများတွင် အသုံးပြုသည့်နည်းပညာဖြစ်သည်။
| ပုံ ၄။ဆိုးဆေးအခြေခံနှင့် စူးစမ်းမှုအခြေခံ qPCR အကြား ကွာခြားချက်များ။ |
Isothermal Amplification
အထက်ဖော်ပြပါ PCR နည်းစနစ်များသည် denaturation၊ annealing နှင့် extension အဆင့်များအတွက် အခန်းအပူချိန်များ အတက်အဆင်း တိကျစွာ အရှိန်မြှင့်ရန် စျေးကြီးသော စက်ဘီးစီးကိရိယာ လိုအပ်ပါသည်။ထိုသို့သောတိကျသောကိရိယာများမလိုအပ်ဘဲ ရိုးရှင်းသောရေချိုးခန်းတွင် သို့မဟုတ် စိတ်ပါဝင်စားသည့်ဆဲလ်များအတွင်း၌ပင် လုပ်ဆောင်နိုင်သော နည်းပညာများစွာကို တီထွင်ထားပါသည်။ဤနည်းပညာများကို isothermal amplification ဟုခေါ်ပြီး ကိန်းဂဏန်း၊ မျဉ်းနား သို့မဟုတ် ကာစကိတ်ချဲ့ခြင်းအပေါ် အခြေခံ၍ အလုပ်လုပ်ပါသည်။
အထင်ရှားဆုံး isothermal amplification အမျိုးအစားမှာ loop-mediated isothermal amplification သို့မဟုတ် LAMP ဖြစ်သည်။LAMP သည် ပုံစံပလိတ် DNA သို့မဟုတ် RNA ကိုချဲ့ထွင်ရန် 65⁰C တွင် exponential amplification ကိုအသုံးပြုသည်။LAMP ကို လုပ်ဆောင်သောအခါ၊ ပစ်မှတ် DNA ၏ ဧရိယာများအတွက် ဖြည့်စွက် primers လေးခုမှ ခြောက်ခုကို DNA အသစ်များ ပေါင်းစပ်ရန်အတွက် DNA polymerase ကို အသုံးပြုပါသည်။ဤ primer နှစ်ခုတွင် အခြားသော primer များတွင် sequences များကို မှတ်မိပြီး ချည်နှောင်ထားသည့် အခမဲ့ sequence များပါရှိပြီး၊ အသစ်ပြုလုပ်ထားသော DNA တွင် "ကွင်း" ဖွဲ့စည်းတည်ဆောက်မှုကို ခွင့်ပြုပေးကာ နောက်ဆက်တွဲချဲ့ထွင်မှုတွင် primer annealing ကို အထောက်အကူဖြစ်စေပါသည်။မီးချောင်းအား fluorescence၊ agarose gel electrophoresis သို့မဟုတ် colorimetry အပါအဝင် နည်းလမ်းများစွာဖြင့် မြင်နိုင်သည်။အရောင်လိုက်တိုင်းတာခြင်းဖြင့် ထုတ်ကုန်၏ရှိနေခြင်း သို့မဟုတ် မရှိခြင်းတို့ကို မြင်သာစေရန်နှင့် အလွယ်တကူသိရှိနိုင်ခြင်းသည် လိုအပ်သောစျေးကြီးသောကိရိယာများမရှိခြင်းကြောင့် LAMP သည် SARS-CoV-2 စမ်းသပ်ခြင်းအတွက် သင့်လျော်သောရွေးချယ်မှုကို ဖြစ်စေသည်၊ သို့မဟုတ် သိုလှောင်မှုနှင့် နမူနာများကို သယ်ဆောင်ခြင်းမပြုနိုင်သောနေရာများတွင် သင့်လျော်သောရွေးချယ်မှုတစ်ခုဖြစ်စေခဲ့သည်။ ဖြစ်နိုင်ချေမရှိပါ၊ သို့မဟုတ် ယခင်က PCR အပူချိန်ထိန်းကိရိယာမပါသော ဓာတ်ခွဲခန်းများတွင်ဖြစ်သည်။
စာတိုက်အချိန်- သြဂုတ်-၁၉-၂၀၂၃