RT-qPCR စမ်းသပ်ချက်တွင် RNA ထုတ်ယူခြင်းနှင့် အရည်အသွေးအကဲဖြတ်ခြင်း၊ ပြောင်းပြန်ကူးယူခြင်း နှင့် qPCR အဆင့်သုံးဆင့်ပါဝင်သည်၊ အဆင့်တစ်ခုစီတွင် ကြိုတင်ကာကွယ်မှုများများစွာပါရှိသည်၊ အောက်တွင်အသေးစိတ်မိတ်ဆက်ပေးပါမည်။
Ⅰ။RNA အရည်အသွေး အကဲဖြတ်ခြင်း။
RT-qPCR စမ်းသပ်မှုတွင်၊ RNA ထုတ်ယူမှုပြီးစီးပြီးနောက်၊ RNA ၏အရည်အသွေးကို အကဲဖြတ်ရန် လိုအပ်ပြီး ၎င်းသည် အရည်အချင်းပြည့်မီမှသာ နောက်ဆက်တွဲစမ်းသပ်မှုကို လုပ်ဆောင်နိုင်မည်ဖြစ်သည်။အကဲဖြတ်ခြင်းနည်းလမ်းများတွင် spectrophotometer၊ Agilent gel electrophoresis၊ Agilent 2100 ခွဲခြမ်းစိတ်ဖြာမှုတို့ ပါဝင်ပြီး အသုံးအများဆုံး spectrophotometer နှင့် agarose gel electrophoresis နည်းလမ်းတို့ကို ထောက်လှမ်းခြင်း တို့ပါဝင်သည်။RNA ၏ အရည်အသွေးကို သေချာစေရန်အတွက် RNA အာရုံစူးစိုက်မှု၊ သန့်ရှင်းမှုနှင့် သမာဓိရှိမှုတို့ကို ရှာဖွေဖော်ထုတ်ခြင်းနှင့် ခွဲခြမ်းစိတ်ဖြာမှု အပြီးသတ်ရန် ဤနည်းလမ်းနှစ်ခုကို အတူတူအသုံးပြုရန် လိုအပ်ကြောင်း သတိပြုသင့်သည်။
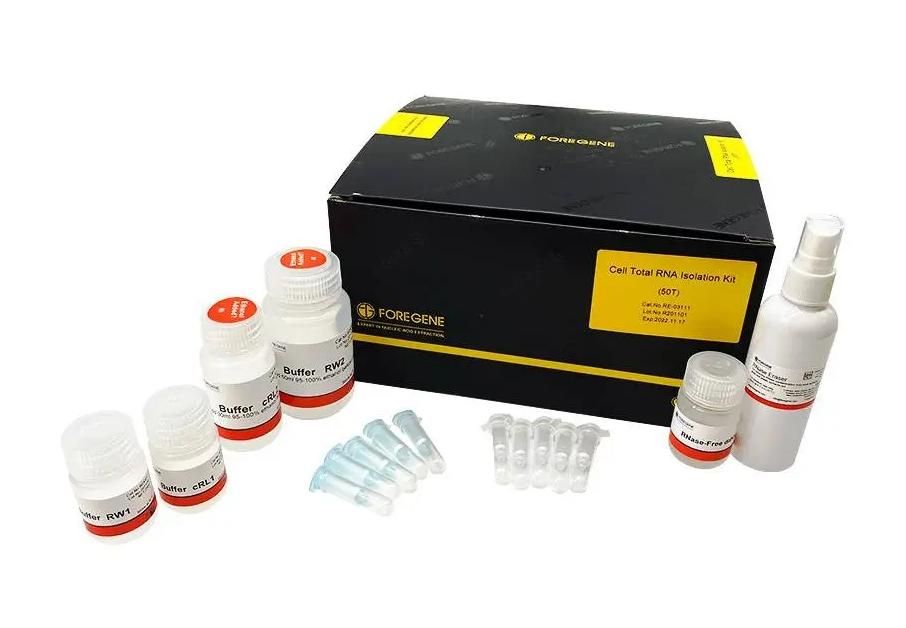
သက်ဆိုင်ရာ RNA Isolation Kit-
ဆဲလ်စုစုပေါင်း RNA သီးခြားခွဲထုတ်ကိရိယာ
သန့်စင်ပြီး အရည်အသွေးမြင့် RNA ကို 11 မိနစ်အတွင်း အမျိုးမျိုးသော ဆဲလ်များမှ ရရှိနိုင်သည်။
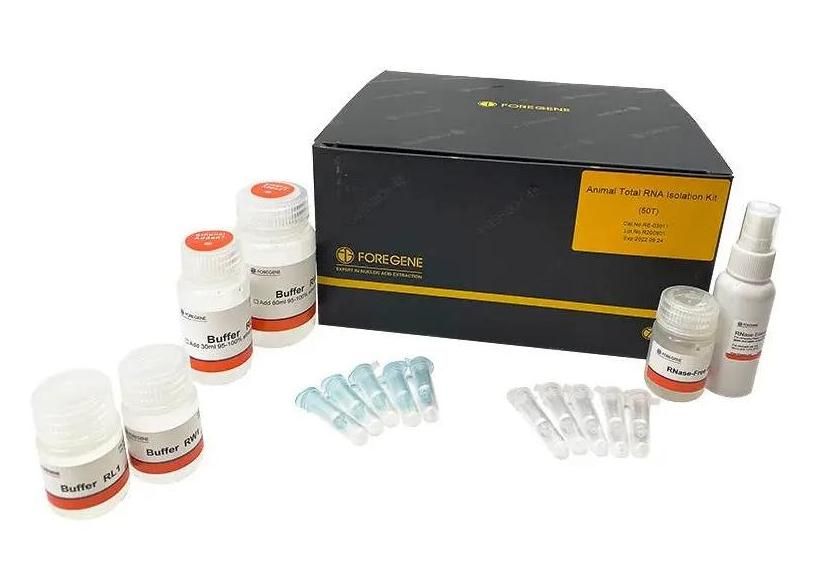
တိရစ္ဆာန်စုစုပေါင်း RNA သီးခြားခွဲထုတ်ကိရိယာ
တိရစ္ဆာန်တစ်ရှူးအမျိုးမျိုးမှ သန့်ရှင်းစင်ကြယ်ပြီး အရည်အသွေးမြင့် စုစုပေါင်း RNA ကို လျင်မြန်ထိရောက်စွာ ထုတ်ယူပါ။
Spectrophotometer-
spectrophotometer ကို အဓိကအားဖြင့် RNA ၏ အာရုံစူးစိုက်မှုနှင့် သန့်ရှင်းစင်ကြယ်မှုကို ဆုံးဖြတ်ရန် အသုံးပြုသော်လည်း RNA နှင့် မျိုးရိုးဗီဇအကြွင်းအကျန်များ၏ သမာဓိကို မတွေ့နိုင်ပါ။၎င်းတို့အနက် A260/280 နှင့် A260/230 တို့သည် RNA သန့်စင်မှုရှာဖွေခြင်းအတွက် အရေးကြီးသောဘောင်များဖြစ်ပြီး ၎င်းတို့၏တန်ဖိုးများအတက်အကျအရ RNA သန့်စင်မှုကို ရှာဖွေတွေ့ရှိနိုင်သည်-
1. 1.9< A260/280< 2.1၊ RNA သန့်စင်မှုသည် ကောင်းမွန်ကြောင်း ညွှန်ပြသည်၊A260/280<1.9၊ RNA တွင် ပရိုတင်းဓာတ်ကျန်ရှိနိုင်သည်ကို ညွှန်ပြသည်၊A260/280>2.1၊ Agarose gel electrophoresis ဖြင့် ထပ်မံအတည်ပြုနိုင်သည့် RNA ၏ တစ်စိတ်တစ်ပိုင်းပြိုကွဲခြင်း ဖြစ်နိုင်ကြောင်း ညွှန်ပြသည်။
2. 2.0< A260/230< 2.2၊ RNA သန့်စင်မှုသည် ကောင်းမွန်ကြောင်း ညွှန်ပြသည်၊A260/230< 2.0 သည် ဖီနော၊ အီသနော သို့မဟုတ် သကြားများကဲ့သို့ RNA တွင် အော်ဂဲနစ်ဓာတ်အကြွင်းအကျန်များ ရှိနိုင်သည်ကို ညွှန်ပြသည်။
Agarose gel electrophoresis:
Agarose gel electrophoresis assay သည် RNA ခိုင်မာမှု၊ ဂျီနိုမ်နှင့် ပရိုတိန်းအကြွင်းအကျန်များကို ပိုင်းခြားစိတ်ဖြာနိုင်သော်လည်း RNA ၏ပြင်းအားကို တိကျစွာတွက်ချက်နိုင်သည် သို့မဟုတ် အော်ဂဲနစ်ဓာတ်ပစ္စည်းများ၏ အကြွင်းအကျန်များကို ရှာဖွေတွေ့ရှိနိုင်မည်မဟုတ်ပေ။ဥပမာအားဖြင့် eukaryotic RNA နမူနာများကို ယူပါ။
1. RNA သည် agarose gel electrophoresis ကို ခံယူထားသည်။ဂျယ်မြေပုံပေါ်တွင် 28sRNA၊ 18sRNA နှင့် 5.8sRNA တစ်ခုတည်းသော band သုံးခုသာရှိခဲ့ပါက၊ ထုတ်ယူထားသော RNA သည် နဂိုအတိုင်းဖြစ်ကြောင်း ညွှန်ပြပါသည်။ဆွဲငင်ခြင်းဖြစ်စဉ်တစ်ခုရှိလျှင် ၎င်းသည် RNA ၏ တစ်စိတ်တစ်ပိုင်း ဆုတ်ယုတ်မှုကို ဖော်ပြသည်။
2. ကော်အပေါက်နှင့် 28sRNA တီးဝိုင်းကြားတွင် တောက်ပသော တီးဝိုင်းတစ်ခု ရှိပါက၊ မျိုးဗီဇ DNA အကြွင်းအကျန်များ ရှိနိုင်ပါသည်။
3. ကော်ပေါက်တွင် ကြိုးများပေါ်လာပါက ပရိုတင်းနှင့် အခြား macromolecular ဒြပ်စင်များ အကြွင်းအကျန်များ ရှိနေနိုင်သည်ကို ညွှန်ပြသည်။
Ⅱ. စာသားပြောင်းပြန်
RNA ထုတ်ယူခြင်းပြီးဆုံးပြီးနောက်၊ ၎င်းအား နောက်ဆက်တွဲစမ်းသပ်မှုများအတွက် cDNA သို့ပြောင်းရန်လိုအပ်သည်၊ ထို့ကြောင့် ပြောင်းပြန်အဆင့်သည် မရှိမဖြစ်လိုအပ်ပါသည်။Reverse transcription ကို reverse transcriptase နှင့် primer ရွေးချယ်မှုမှ မိတ်ဆက်ပေးပါမည်-
ပြောင်းပြန် စာသားမှတ်တမ်း ရွေးချယ်မှု-
ပုံမှန် ပြောင်းပြန် စာသားမှတ်တမ်းများတွင် AMV RTase နှင့် MMLV RTase ပါဝင်သည်။AMV RTase ၏ RNase H သည် အားကောင်းသော လုပ်ဆောင်ချက်၊ တိုတောင်းသော ပေါင်းစပ်မှု အရှည်၊ ပေါင်းစပ်မှု ပမာဏ နည်းပါးပြီး ကောင်းသော အပူတည်ငြိမ်မှု (42 ~ 55 ℃) ရှိသည်။MMLV RTase ၏ RNase H လုပ်ဆောင်ချက် အားနည်းသည်၊ ပေါင်းစပ်မှု အရှည်သည် ရှည်လျားသည်၊ ပေါင်းစပ်မှု ပမာဏ မြင့်မားသည်၊ အပူတည်ငြိမ်မှု ညံ့သည် (37 ~ 42 ℃)။
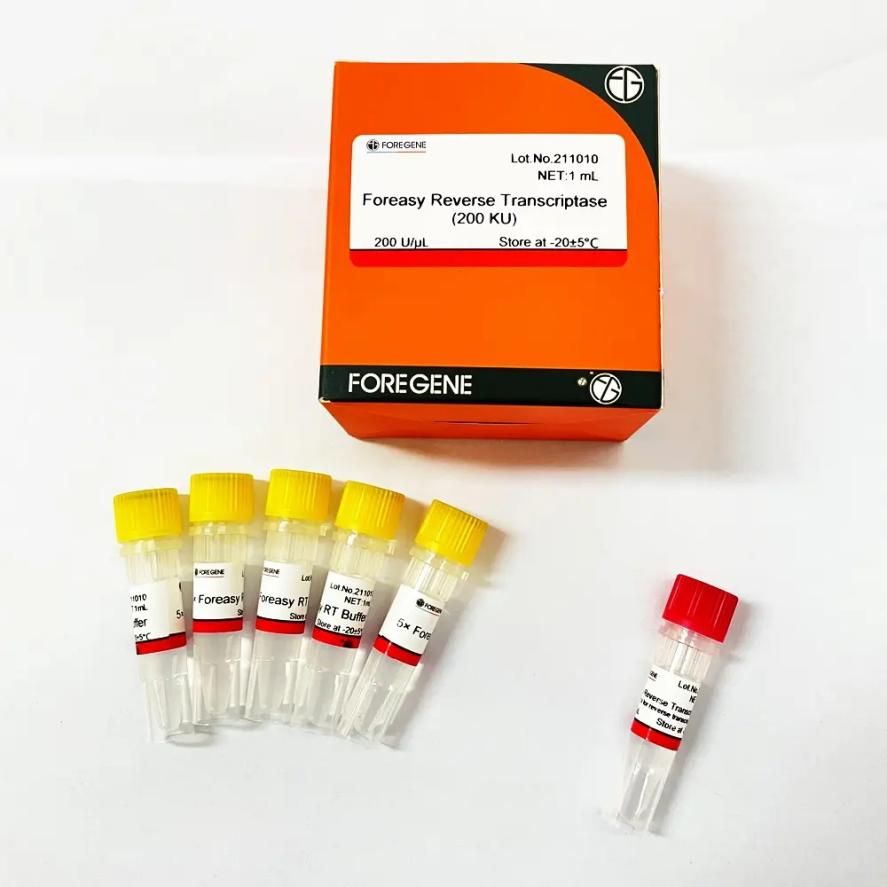
RNase H အင်ဇိုင်းသည် RNA ပုံစံပလိတ်ကို ပျက်စီးစေသော လုပ်ဆောင်မှုဖြစ်သောကြောင့်၊ အားနည်းသော RNase H လုပ်ဆောင်ချက်ပါရှိသော MMLV အား ပြောင်းပြန်ကူးယူခြင်းတွင် ဦးစားပေးရွေးချယ်သင့်ပြီး နောက်ပိုင်းတွင် မျိုးရိုးဗီဇအင်ဂျင်နီယာချုပ်ပြီးနောက်၊ MMLV ၏ အပူပိုင်းတည်ငြိမ်မှုသည် အရည်အသွေးကောင်းမွန်သော ခုန်ပျံကျော်လွှားမှုသို့ ရောက်ရှိသွားပါသည်။Foregene ယူခြင်း။Foreasy Reverse Transcriptase(M-MLV ပြောင်းပြန်ကူးယူခြင်းအတွက်) ဥပမာအနေဖြင့်၊ ၎င်းသည် မျိုးရိုးဗီဇပြန်လည်ပေါင်းစပ်ခြင်းနည်းပညာကို အသုံးပြု၍ E. coli အင်ဂျင်နီယာဘက်တီးရီးယားတွင် ဖော်ပြထားသော ပြောင်းပြန် transcriptase အသစ်ဖြစ်သည်။၎င်းသည် single-stranded RNA၊ DNA သို့မဟုတ် RNA:DNA hybrid မှ ဖြည့်စွက် DNA ကြိုးကို ပေါင်းစပ်ပေါင်းစပ်ထားသော DNA polymerase တစ်ခုဖြစ်သည်။၎င်းတွင် RNase H လုပ်ဆောင်ချက်၊ ခိုင်ခံ့သော တည်ငြိမ်မှု၊ ခိုင်ခံ့သော RNA ရင်းနှီးမှုနှင့် မြင့်မားသော အာရုံခံနိုင်စွမ်းတို့ မရှိပါ။
Foreasy Reverse Transcriptase(M-MLV ပြောင်းပြန်ကူးယူခြင်းအတွက်)
primer ရွေးချယ်မှု
ယေဘုယျအားဖြင့် RT primers များသည် oligo dT၊ ကျပန်း primers နှင့် gene-specific primers ဟူ၍ အမျိုးအစားသုံးမျိုးရှိသည်။မတူညီသော စမ်းသပ်မှုလိုအပ်ချက်များအရ အသုံးပြုရန် သင့်လျော်သော primers ကိုရွေးချယ်ပါ။
1. နမူနာပုံစံသည် eukaryotic မူရင်းဖြစ်ပြီး နောက်ကျသော cDNA ကို ပုံမှန် PCR ချဲ့ထွင်ရန်အတွက် အသုံးပြုပါက၊ Oligo (dT) ကို အကြံပြုထားသည်။နောက်ဆက်တွဲစမ်းသပ်ချက်ကို qPCR အတွက်သာအသုံးပြုပါက၊ ပြောင်းပြန်ကူးယူခြင်း၏ထိရောက်မှုကိုတိုးတက်စေရန် Oligo (dT) ကို ကျပန်း primers များနှင့် ရောစပ်ရန် အကြံပြုထားသည်။
2. နမူနာပုံစံသည် prokaryotes မှဖြစ်ပါက၊ Random Primers သို့မဟုတ် gene သီးသန့် primer များကို reverse transcription အတွက် ရွေးချယ်သင့်သည်။
Ⅲ.qPCR
Fluorescence quantification ကို quantitative method ရွေးချယ်မှု၊ primer design စည်းမျဉ်းများ၊ ROX ရွေးချယ်မှု၊ တုံ့ပြန်မှုစနစ်ဖွဲ့စည်းပုံနှင့် တုံ့ပြန်မှုအခြေအနေများ သတ်မှတ်ခြင်းစသည်ဖြင့် အဓိကအားဖြင့် အသေးစိတ်ဖော်ပြထားပါသည်။
အရေအတွက်နည်းများ ရွေးချယ်ခြင်း-
အရေအတွက်နည်းများကို နှိုင်းယှဥ်အရေအတွက်နည်းများ နှင့် အကြွင်းမဲ့အရေအတွက်နည်းများ ဟူ၍ ပိုင်းခြားထားသည်။မျိုးရိုးဗီဇဖော်ပြမှုအပေါ် အချို့သော ကုသမှုနည်းလမ်းများ၏ အကျိုးသက်ရောက်မှုကို ရှာဖွေရန်၊ မတူညီသောအချိန်များတွင် မျိုးရိုးဗီဇဖော်ပြမှုကွာခြားချက်ကို ရှာဖွေရန်နှင့် မတူညီသောတစ်ရှူးများတွင် ဗီဇဖော်ပြမှုကွာခြားချက်ကို နှိုင်းယှဉ်ရန် နှိုင်းရအရေအတွက်ကို အသုံးပြုနိုင်သည်။အကြွင်းမဲ့ ပမာဏသည် ဗိုင်းရပ်စ်ရှိ နူကလိစ်အက်ဆစ် ပမာဏကို သိရှိနိုင်သည် ။စမ်းသပ်မှုများပြုလုပ်သောအခါ၊ ကျွန်ုပ်တို့သည် ကျွန်ုပ်တို့၏ကိုယ်ပိုင်စမ်းသပ်မှုများအရ သင့်လျော်သော အရေအတွက်နည်းလမ်းများကို ရွေးချယ်ရမည်ဖြစ်သည်။
Primer ဒီဇိုင်းအခြေခံမူများ
qPCR အတွက် primer ဒီဇိုင်းသည် ချဲ့ထွင်မှု ထိရောက်မှုနှင့် ထုတ်ကုန်တိကျမှုတို့နှင့် တိုက်ရိုက်သက်ဆိုင်ပါသည်။ထို့ကြောင့်၊ ကောင်းမွန်သော primers များကို မှန်ကန်စွာ ဒီဇိုင်းဆွဲခြင်းသည် အောင်မြင်သော qPCR ၏ ပထမခြေလှမ်းဖြစ်သည်။primer ဒီဇိုင်းတွင် သမားရိုးကျ primer ဒီဇိုင်းနိယာမကို လိုက်နာသောအခါတွင် အောက်ပါအခြေခံမူများကို ဂရုပြုသင့်သည်-
1. ပစ်မှတ်အပိုင်းအစ၏ အရှည်ကို 100 နှင့် 300 bp အကြား ထိန်းချုပ်ထားသည်။
2. မျိုးဗီဇ DNA လွှမ်းမိုးမှုကို ရှောင်ရှားရန် Cross-exon ဒီဇိုင်း၊
3. ဒီဇိုင်းထုတ်ထားသော primers များကို amplification efficiency အတွက် စမ်းသပ်ရန် လိုအပ်ပြီး amplification efficiency စံ (90-110%) သို့ရောက်ရှိမှသာ quantitative tests များအတွက် ၎င်းတို့ကို အသုံးပြုနိုင်ပါသည်။
4. Primer အာရုံစူးစိုက်မှုကို အများအားဖြင့် 0.1uM နှင့် 1.0uM ကြားတွင် အကောင်းဆုံးဖြစ်အောင် ပြုလုပ်ထားသည်။
ရွေးချယ်မှုROX-
ပမာဏတုံ့ပြန်မှုဖြစ်စဉ်တွင် ROX သည် အငွေ့ပျံခြင်းနှင့် ငွေ့ရည်ဖွဲ့ခြင်းကြောင့် ဖြစ်ပေါ်လာသော ထုထည်ခြားနားချက်၊ အလင်းလမ်းကြောင်းခြားနားချက်ကို ချိန်ညှိနိုင်ပြီး ရလဒ်များ၏ ထပ်တလဲလဲဖြစ်နိုင်မှုကို ပိုမိုကောင်းမွန်စေသည်။သို့သော် ROX ရွေးချယ်မှုသည် တူရိယာနှင့် သက်ဆိုင်ကြောင်း သတိပြုသင့်သည်။qPCR တူရိယာတွင် အပေါက်များကြား ခြားနားချက်ကို အလိုအလျောက် ပြုပြင်ပေးသည့် လုပ်ဆောင်ချက် ပါရှိပါက ROX ထည့်ရန် မလိုအပ်ပါ။မဟုတ်ရင် ROX ပြုပြင်မှု ထည့်ဖို့ လိုပါတယ်။ဓါတ်ကူပစ္စည်းများဝယ်ယူရာတွင် အသေးစားလုပ်ဖော်ကိုင်ဖက်များသည် မှန်ကန်သော ROX ကိုရွေးချယ်ရန်အတွက် အသုံးပြုသည့်ကိရိယာအတိုင်းဖြစ်ရမည်၊ နောက်ပိုင်းအမှားများကိုရှောင်ရှားပါ။
တုံ့ပြန်မှုစနစ်ပြင်ဆင်မှု
20ul နှင့် 50ul တို့၏ တုံ့ပြန်မှုပမာဏကို ဦးစားပေးသည်။စနစ်ကို ရေးဆွဲသည့်အခါတွင် အောက်ပါကိစ္စရပ်များကို ဂရုပြုသင့်သည်။
1. အလွန်သန့်ရှင်းသော အလုပ်ခုံတန်းလျား၊ ddH အသစ်တွင် လေဝင်လေထွက်ဖြင့် တုံ့ပြန်မှုစနစ်အား ပြင်ဆင်ထားရန် လိုအပ်ပါသည်။2စမ်းသပ်မှုတစ်ခုစီအတွက် O ကို အသုံးပြုသည်။
2. စမ်းသပ်မှုတစ်ခုစီသည် စနစ်အတွင်း လေထုညစ်ညမ်းမှုရှိမရှိ စစ်ဆေးရန် NTC ကို ပြင်ဆင်ရန် လိုအပ်ပြီး စနစ်ပြင်ဆင်သည့်အခါတွင် primer တစ်စုံစီသည် NTC ပြုလုပ်ရန် လိုအပ်ပါသည်။
3. RNA နမူနာပုံစံတွင် gDNA အကြွင်းအကျန်ရှိမရှိကို စစ်ဆေးရန်၊ ထောက်လှမ်းရန်အတွက် နမူနာတစ်ခုစီအတွက် NRT ကို ပြင်ဆင်နိုင်သည်။
4. စနစ်အား ပြင်ဆင်သည့်အခါ နမူနာတစ်ခုအတွက် အနည်းဆုံး နည်းပညာဆိုင်ရာ ထပ်ခါထပ်ခါ 3 ကြိမ်ပြုလုပ်ရန် အကြံပြုထားသည်။
5. နမူနာပုံစံသည် cDNA ဖြစ်သောအခါ၊ qPCR စမ်းသပ်မှုတွင် ပြောင်းပြန်ကူးယူခြင်းစနစ်၏ ဟန့်တားမှုအကျိုးသက်ရောက်မှုကို လျှော့ချရန် 5-10 ကြိမ်မှေးမှိန်ရန် အကြံပြုထားသည်။CT တန်ဖိုးသည် 20-30 ကြားတွင် ကျဆင်းစေရန် ပုံစံခွက်ပမာဏကို gradient ဖြင့် စူးစမ်းခြင်းက ပိုကောင်းပါသည်။
6. လိုအပ်သောတုံ့ပြန်မှုအရေအတွက်ကိုဆုံးဖြတ်ပါ၊ တုံ့ပြန်မှုအရေအတွက်၏အခြေခံပေါ်တွင် 5-10% တိုးကာ ထုထည်ဖွဲ့စည်းမှုနံပါတ်ကို တွက်ချက်ပါ။
7၊ စနစ်သည် premix နိယာမကိုအသုံးပြု၍ centrifugation ပြီးနောက်ရောစပ်ပြီးပူဖောင်းများမရှိစေရန်သေချာသည်။
8၊ တတ်နိုင်သမျှ ပံ့ပိုးပေးသော စားသုံးကုန်များကို ရွေးချယ်ပါ။
ဆက်စပ် RT-qPCR Kit
ပစ္စည်းကိရိယာသည် ထူးခြားသော Foregene ပြောင်းပြန် စာသားပြောင်းပြန်ဓါတ်ဆားနှင့် Foregene HotStar Taq DNA Polymerase နှင့် တုံ့ပြန်မှု၏ ချဲ့ထွင်မှု၏ ထိရောက်မှုနှင့် တိကျမှုတို့ကို ထိထိရောက်ရောက် မြှင့်တင်ရန် တုံ့ပြန်မှုစနစ်အား ထိထိရောက်ရောက် မြှင့်တင်ပေးပါသည်။
ပို့စ်အချိန်- ဧပြီလ ၂၃-၂၀၂၃