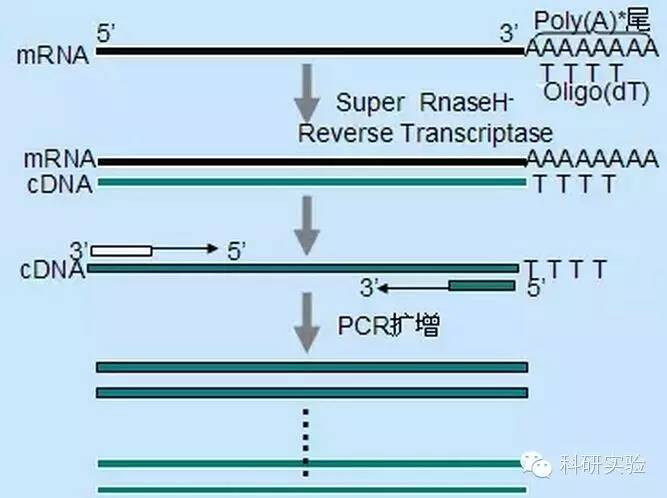
Ⅰ. တုံ့ပြန်မှုစနစ်၏ အာရုံခံနိုင်စွမ်းကို တိုးစေသည်။
1. အရည်အသွေးမြင့် RNA ကို ခွဲခြားထားသည်-
အောင်မြင်သော cDNA ပေါင်းစပ်မှုသည် အရည်အသွေးမြင့် RNA မှလာသည်။အရည်အသွေးမြင့် RNA သည် အနည်းဆုံး စုစုပေါင်းကြာရှည်ကြောင်း သေချာစေသင့်ပြီး EDTA သို့မဟုတ် SDS ကဲ့သို့သော အသံသွင်းအင်ဇိုင်းများ မပါဝင်သည့် inhibitors များ မပါဝင်ပါ။RNA ၏ အရည်အသွေးသည် သင် cDNA သို့ ကူးယူနိုင်သော ဆက်တိုက်အချက်အလက်၏ အမြင့်ဆုံးတန်ဖိုးကို ဆုံးဖြတ်သည်။ယေဘူယျအားဖြင့် RNA သန့်စင်ခြင်းနည်းလမ်းသည် isooocyanate/acidophenol ကိုအသုံးပြုသည့် အဆင့်နည်းလမ်းဖြစ်သည်။RNase ၏ညစ်ညမ်းမှုကိုကာကွယ်ရန်အတွက်၊ RNase ကြွယ်ဝသောနမူနာမှခွဲထုတ်ထားသော RNA (ပန်ကရိယကဲ့သို့သော) အရည်အသွေးမြင့် RNA ကိုကယ်တင်ရန် Formaldehyde သိုလှောင်မှုလိုအပ်သည်၊ ၎င်းသည်ရေရှည်သိုလှောင်မှုအတွက်ပိုကောင်းသည်။ကြွက်အသည်းမှ ထုတ်ယူရရှိသော RNA သည် ရေတွင် တစ်ပတ်ကြာ သိုလှောင်ပြီးနောက် အခြေခံအားဖြင့် ဆုတ်ယုတ်သွားခဲ့ပြီး ကြွက်သရက်ရွက်မှ ထုတ်ယူသော RNA သည် ရေထဲတွင် သုံးနှစ်ကြာ သိုလှောင်ပြီးနောက် တည်ငြိမ်နေပါသည်။ထို့အပြင်၊ 4kb ထက်ကြီးသော စာသားများသည် သေးငယ်သော စာသားများထက် RNase ဆုတ်ယုတ်မှုကို ခြေရာခံရန် ပို၍ အကဲဆတ်ပါသည်။သိုလှောင်မှု RNA နမူနာ၏ တည်ငြိမ်မှုကို တိုးမြှင့်ရန်အတွက် RNA ကို မက်သလမင်း အိုင်းယွန်းတစ်ခုတွင် ပျော်ဝင်နိုင်ပြီး -70°C သိမ်းဆည်းထားသည်။RNA ကို သိမ်းဆည်းရာတွင် အသုံးပြုသော Thylide သည် RNA ကို ပျက်ပြားစေသော အထွေထွေ အရာဝတ္ထု မပါဝင်ရပါ။ပန်ကရိယမှဆင်းသက်လာသော RNA ကို မက်သလမင်းတွင် အနည်းဆုံး တစ်နှစ်အထိ သိမ်းဆည်းနိုင်သည်။RNA ကို အသုံးပြုရန် အဆင်သင့်ဖြစ်သောအခါတွင်၊ RNA ကို စုပ်ယူရန် အောက်ပါနည်းလမ်းများကို အသုံးပြုနိုင်သည်- NaCl ကို 0.2m နှင့် အီသနော၏ ထုထည် 4 ဆ ပေါင်းထည့်ကာ အခန်းအပူချိန်ကို 3-5 မိနစ်ကြာ ထားကာ 10,000 × g centrifugal ကို 5 မိနစ်ကြာ ထားလိုက်ပါ။
2. RNaseH လုပ်ဆောင်ချက် (RNaseH-) မပါဘဲ ပြောင်းပြန် စာသားမှတ်တမ်းကို အသုံးပြုပါ။
RNase inhibitors များကို cDNA ပေါင်းစပ်မှု၏ အရှည်နှင့် အထွက်နှုန်းကို တိုးမြင့်ရန်အတွက် ပြောင်းပြန် ကူးယူတုံ့ပြန်မှုများတွင် မကြာခဏ ပေါင်းထည့်ပါသည်။RNase inhibitor ကို buffers များရှေ့မှောက်တွင် နှင့် DTT ကဲ့သို့သော လျှော့ချအေးဂျင့်များထံတွင် RNase inhibitor ကို ပေါင်းထည့်ထားပြီး RNA ကို ကျဆင်းစေသော ချည်နှောင်ထားသော RNases များကို ထုတ်ပေးသောကြောင့်၊ပရိုတိန်း RNase inhibitor သည် RNase A, B, C အားဖြင့် RNA ၏ပြိုကွဲမှုကိုသာကာကွယ်ပေးပြီး အရေပြားပေါ်ရှိ RNases များကိုမကာကွယ်နိုင်သောကြောင့် အဆိုပါ inhibitors များကိုအသုံးပြုနေသော်လည်း လက်ချောင်းများမှ RNases မိတ်ဆက်ခြင်းမပြုရန် သတိပြုသင့်သည်။
Reverse transcriptase သည် RNA ၏ cDNA အဖြစ်သို့ ပြောင်းလဲခြင်းကို လှုံ့ဆော်ပေးသည်။M-MLV နှင့် AMV နှစ်မျိုးလုံးတွင် ၎င်းတို့၏ကိုယ်ပိုင် polymerase လုပ်ဆောင်မှုအပြင် endogenous RNaseH လုပ်ဆောင်ချက် ရှိသည်။RNaseH လုပ်ဆောင်ချက်သည် RNA နမူနာများနှင့် DNA ပရိုဂရမ်များ သို့မဟုတ် cDNA တိုးချဲ့ကြိုးများကြားတွင် ပေါင်းစပ်ဖွဲ့စည်းထားသော ဟေတီရိုဇိုင်းဂစ်ကြိုးများအတွက် polymerase လုပ်ဆောင်ချက်နှင့် ယှဉ်ပြိုင်ပြီး DNA ရှုပ်ထွေးမှုများရှိ RNA ကြိုးများကို ကျဆင်းစေသည်။RNaseH လုပ်ဆောင်ချက်ကြောင့် ပျက်စီးသွားသော RNA နမူနာများကို cDNA ပေါင်းစပ်မှုအတွက် ထိရောက်သော အလွှာများအဖြစ် အသုံးမပြုနိုင်တော့ဘဲ cDNA ပေါင်းစပ်မှု၏ အထွက်နှုန်းနှင့် အရှည်ကို လျှော့ချပေးသည်။ထို့ကြောင့် reverse transcriptase ၏ RNaseH လုပ်ဆောင်ချက်ကို ဖယ်ရှားခြင်း သို့မဟုတ် လျှော့ချခြင်းသည် များစွာအကျိုးရှိမည်ဖြစ်သည်။
SuperScriptⅡ ပြောင်းပြန် စာသားမှတ်တမ်း၊ MMLV RNaseH- နှင့် thermoScript ပြောင်းပြန် စာသားမှတ်တမ်း၊ MMLV ၏ ပြောင်းပြန် စာသားမှတ်တမ်း၊ RNaseH ၏ AMV- သည် MMLV နှင့် AMV ထက် အပြည့်အစုံ cDNA ကို ပိုထုတ်ပေးသည်။RT-PCR အာရုံခံနိုင်စွမ်းသည် cDNA ပေါင်းစပ်ထားသောပမာဏကြောင့် ထိခိုက်သည်။ThermoScript သည် AMV ထက် များစွာပို၍ ထိခိုက်လွယ်သည်။RT-PCR ထုတ်ကုန်များ၏ အရွယ်အစားသည် အထူးသဖြင့် ပိုကြီးသော Cdnas ကို ပုံတူပွားသည့်အခါတွင် cDNA ကို ပြောင်းပြန် transcriptase ၏ စွမ်းရည်ဖြင့် ကန့်သတ်ထားသည်။MMLV နှင့် နှိုင်းယှဉ်ပါက SuperScripⅡသည် ရှည်လျားသော RT-PCR ထုတ်ကုန်များ၏ အထွက်နှုန်းကို သိသိသာသာ တိုးမြင့်စေသည်။RNaseH- ၏ပြောင်းပြန် transcriptase သည် အပူတည်ငြိမ်မှုကိုလည်း တိုးမြင့်စေသောကြောင့် တုံ့ပြန်မှုကို ပုံမှန် 37-42 ℃ထက်မြင့်သော အပူချိန်တွင် လုပ်ဆောင်နိုင်သည်။အကြံပြုထားသောပေါင်းစပ်မှုအခြေအနေများအောက်တွင် oligo(dT) primers နှင့် 10μCi [alpha-p]dCTP ကိုအသုံးပြုခဲ့သည်။ပထမကွင်းဆက်၏ စုစုပေါင်းထုတ်လုပ်မှုကို TCA မိုးရွာသွန်းမှုနည်းလမ်းဖြင့် တွက်ချက်ခဲ့သည်။cDNA အရှည်ကို အရွယ်အစား-စီထားသော အမြှေးပါးဖယ်ရှားခြင်းနှင့် အယ်ကာလိုင်း agarose ဂျယ်ဖြင့် ရေတွက်ခြင်းတို့ကို အသုံးပြု၍ ပိုင်းခြားစိတ်ဖြာထားပါသည်။
3. ပြောင်းပြန်ကူးယူဖော်ပြခြင်း၏ အပူထိန်းသိမ်းမှုအပူချိန်ကို တိုးမြှင့်ပါ-
မြင့်မားသောအပူချိန်သည် RNA ၏ဒုတိယဖွဲ့စည်းပုံကိုဖွင့်ရန်နှင့်တုံ့ပြန်မှု၏အထွက်နှုန်းကိုတိုးစေသည်။RNA ပုံစံပလိတ်အများစုအတွက်၊ RNA နှင့် primer ကို 65°C တွင် ကြားခံ သို့မဟုတ် ဆားမပါဘဲ ကိုင်ထားပြီး ရေခဲပေါ်တွင် လျှင်မြန်စွာ အအေးခံခြင်းဖြင့် ဒုတိယဖွဲ့စည်းပုံအများစုကို ဖယ်ရှားပေးပြီး primer များကို စည်းနိုင်စေပါသည်။သို့သော်၊ အချို့သောပုံစံများသည် အပူလွန်ကဲပြီးနောက်တွင်ပင် ဒုတိယဖွဲ့စည်းပုံရှိသေးသည်။ဤခက်ခဲသောပုံစံများကို ချဲ့ထွင်ခြင်းအား ThermoScript reverse transcriptase သုံးပြီး အသံချဲ့ထွင်ခြင်းအား ပိုမိုကောင်းမွန်စေရန် အပူချိန်မြင့်မြင့်တွင် ပြောင်းပြန် transcriptase တုံ့ပြန်မှုကို ထားရှိခြင်းဖြင့် လုပ်ဆောင်နိုင်ပါသည်။မြင့်မားသောအပူချိန်များသည် gene-specific primers (GSPS) ကို အသုံးပြု၍ အထူးသဖြင့် cDNA ပေါင်းစပ်မှုကို လုပ်ဆောင်သောအခါတွင် တိကျမှုကိုလည်း တိုးလာစေသည် (အခန်း 3 ကိုကြည့်ပါ)။GSP ကို အသုံးပြုပါက၊ primer ၏ Tm တန်ဖိုးသည် မျှော်လင့်ထားသည့် အပူချိန်နှင့် တူညီကြောင်း သေချာပါစေ။60 ℃ အထက် oligo (dT) နှင့် ကျပန်း primer များကို အသုံးမပြုပါနှင့်။ကျပန်း Primer များကို 25 ℃ အပူချိန် 60 ဒီဂရီအထိမတိုးမီ 10 မိနစ်ကြာထိန်းထားရန်လိုအပ်သည်။ပိုမိုမြင့်မားသော ပြောင်းပြန်စာသားမှတ်တမ်းအပူချိန်များကိုအသုံးပြုခြင်းအပြင်၊ RNA/ primer အရောအနှောကို 65 ℃ denaturing အပူချိန်မှ ပြောင်းပြန်စာသားမှတ်တမ်းကိုင်ဆောင်ထားသောအပူချိန်သို့ တိုက်ရိုက်လွှဲပြောင်းပေးပြီး ကြိုတင်အပူပေးထားသော 2× တုံ့ပြန်မှုအရောအနှောကို ပေါင်းထည့်ခြင်းဖြင့် တိကျမှုကို မြှင့်တင်နိုင်ပါသည်။ဤချဉ်းကပ်နည်းသည် အပူချိန်နိမ့်သည့်အချိန်တွင် ဖြစ်ပေါ်သော intermolecular base pairing ကို တားဆီးပေးသည်။PCR တူရိယာကိုအသုံးပြုခြင်းသည် RT-PCR အတွက် လိုအပ်သော အပူချိန်ခလုတ်များစွာကို ရိုးရှင်းစေသည်။
Tth အပူတည်ငြိမ်အောင်ပြုလုပ်ထားသော ပေါ်လီမာရတ်သည် Mn2+ တွင် Mg2+ နှင့် RNA ပေါ်လီမာရစ်၏ရှေ့မှောက်တွင် DNA ပေါ်လီမာရစ်အဖြစ် လုပ်ဆောင်သည်။အပူချိန် 65 ဒီဂရီအထိ ထိန်းထားနိုင်ပါတယ်။သို့ရာတွင်၊ PCR အတွင်း Mn2+ ပါဝင်မှုသည် သစ္စာရှိမှုကို လျော့နည်းစေပြီး၊ ၎င်းသည် cDNA ပုံတူပွားခြင်းကဲ့သို့သော မြင့်မားသောတိကျသောချဲ့ထွင်မှုအတွက် သင့်လျော်မှုနည်းပါးစေသည်။ထို့အပြင်၊ Tth သည် အာရုံခံနိုင်စွမ်းကို လျော့နည်းစေသည့် ပြောင်းပြန်ကူးယူခြင်းတွင် ထိရောက်မှုနည်းပါးပြီး အင်ဇိုင်းတစ်ခုသည် ပြောင်းပြန်ကူးယူခြင်းနှင့် PCR လုပ်ဆောင်နိုင်သောကြောင့်၊ ညစ်ညမ်းသောမျိုးရိုးဗီဇ DNA နှင့် cDNA ၏ ချဲ့ထွင်ထားသောထုတ်ကုန်များကို ခွဲခြားရန် တုံ့ပြန်မှုများကို အသုံးမပြုနိုင်ပါ။
4. ပြောင်းပြန်ကူးယူခြင်းကို အားပေးသည့် ပေါင်းစည်းခြင်း-
glycerin နှင့် DMSO အပါအဝင် additives များကို ပထမကွင်းဆက်ပေါင်းစပ်မှုတုံ့ပြန်မှုသို့ ပေါင်းထည့်ခြင်းသည် nucleic acid double strand ၏တည်ငြိမ်မှုကို လျှော့ချနိုင်ပြီး RNA ဒုတိယဖွဲ့စည်းပုံကို ဖြေလျှော့နိုင်သည်။SuperScriptⅡ သို့မဟုတ် MMLV ၏ လုပ်ဆောင်ချက်များကို မထိခိုက်စေဘဲ 20% glycerin သို့မဟုတ် 10% DMSO အထိ ထည့်နိုင်သည်။AMV သည် လှုပ်ရှားမှုကို မလျှော့ချဘဲ glycerol 20% အထိ သည်းခံနိုင်သည်။SuperScriptⅡ reverse transcription တုံ့ပြန်မှုတွင် RT-PCR ၏ အာရုံခံနိုင်စွမ်းကို အမြင့်ဆုံးမြှင့်တင်ရန် 10% glycerol ကို 45 ℃ တွင် ထည့်သွင်းပြီး လျှပ်ကာလုပ်နိုင်ပါသည်။retrotranscription-reaction ထုတ်ကုန်၏ 1/10 ကို PCR သို့ပေါင်းထည့်ပါက၊ ချဲ့ထွင်သည့်တုံ့ပြန်မှုတွင် glycerol ၏အာရုံစူးစိုက်မှုသည် PCR ကိုတားဆီးရန်မလုံလောက်ပါ။
5. RNaseH လုပ်ဆောင်ခြင်း-
PCR မတိုင်မီ RNaseH နှင့် cDNA ပေါင်းစပ်တုံ့ပြန်မှုများကို ကုသခြင်းဖြင့် အာရုံခံနိုင်စွမ်းကို မြှင့်တင်နိုင်သည်။အချို့သောပုံစံများအတွက်၊ cDNA ပေါင်းစပ်မှုတုံ့ပြန်မှုတွင် RNA သည် ချဲ့ထွင်ထားသောထုတ်ကုန်များ၏ ပေါင်းစပ်မှုကို ဟန့်တားပေးသည်၊ ယင်းအခြေအနေတွင် RNaseH ကုသမှုသည် အာရုံခံနိုင်စွမ်းကိုတိုးမြင့်စေသည်ဟု ယူဆပါသည်။ယေဘူယျအားဖြင့်၊ ကူးယူမှုနည်းပါးသော tuberous scherosisⅡ ကဲ့သို့သော ရှည်လျားသော cDNA ပစ်မှတ်ပုံစံပုံစံကို ချဲ့ထွင်ရန်အတွက် RNaseH ကုသမှု လိုအပ်ပါသည်။ဤခက်ခဲသောပုံစံအတွက်၊ RNaseH သည် SuperScriptⅡ သို့မဟုတ် AMV ဖြင့်ပေါင်းစပ်ထားသော cDNA မှထုတ်ပေးသောအချက်ပြမှုကို မြှင့်တင်ပေးပါသည်။RT-PCR တုံ့ပြန်မှုအများစုအတွက်၊ RNaseH ကုသမှုသည် 95 ℃ insulated PCR denaturation အဆင့်သည် ပုံမှန်အားဖြင့် RNA မှ RNA ကို hydrolyzes ဖြစ်သောကြောင့် RNaseH ကုသမှုသည် ရွေးချယ်နိုင်သောကြောင့်ဖြစ်သည်။
6. RNA ပမာဏအနည်းငယ်ကို ရှာဖွေတွေ့ရှိရန် ပိုမိုကောင်းမွန်သောနည်းလမ်းများ
RNA အနည်းငယ်သာရရှိနိုင်သောအခါ RT-PCR သည် အထူးသဖြင့် စိန်ခေါ်မှုဖြစ်သည်။RNA ခွဲခြားမှုအတွင်း glycogen ၏သယ်ဆောင်သူအဖြစ်ထပ်ပေါင်းခြင်းသည်နမူနာငယ်များ၏အထွက်နှုန်းကိုတိုးစေသည်။Trizol ကဲ့သို့ RNase-free glycogen ကို တစ်ချိန်တည်းတွင် ထည့်နိုင်သည်။Glycogen သည် ရေတွင်ပျော်ဝင်နိုင်ပြီး နောက်ဆက်တွဲမိုးရွာသွန်းမှုတွင် ကူညီပေးရန်အတွက် RNA နှင့်အတူ ရေအဆင့်တွင်ရှိနေနိုင်သည်။RNase-free glycogen ၏ အကြံပြုထားသော အာရုံစူးစိုက်မှုသည် တစ်ရှူး 50mg သို့မဟုတ် 106 ဆဲလ်များထက်နည်းသော နမူနာများအတွက် 250μg/ml ဖြစ်သည်။
SuperScriptⅡကို အသုံးပြု၍ စာသားပြောင်းပြန်လှန်တုံ့ပြန်မှုများအတွက် acetylated BSA ၏ထပ်တိုးမှုသည် အာရုံခံနိုင်စွမ်းကို တိုးစေပြီး RNA ပမာဏအနည်းငယ်အတွက် SuperScriptⅡ ပမာဏကို လျှော့ချကာ RnaseOut nuclease inhibitor ၏ ယူနစ် 40 ပေါင်းထည့်ခြင်းဖြင့် ထောက်လှမ်းမှုအဆင့်ကို မြှင့်တင်နိုင်ပါသည်။RNA ခွဲခြားရာတွင် glycogen ကိုအသုံးပြုပါက၊ SuperScriptⅡကိုအသုံးပြု၍ စာသားမှတ်တမ်းတုံ့ပြန်မှုများကိုပြောင်းပြန်ဖြစ်စေရန်အတွက် BSA သို့မဟုတ် RNase inhibitors များထပ်ဖြည့်ခြင်းကို အကြံပြုထားပါသည်။
Ⅱ. RT-PCR ၏ တိကျမှုကို မြှင့်တင်ပါ။
1. cNDA ပေါင်းစပ်မှု-
ပထမကြိုးမျှင် cDNA ပေါင်းစပ်မှုကို အစပြုရန် မတူညီသော နည်းလမ်းသုံးခုကို အသုံးပြုနိုင်ပြီး နည်းလမ်းတစ်ခုစီ၏ နှိုင်းယှဥ်တိကျမှုသည် cDNA ပေါင်းစပ်ထားသော ပမာဏနှင့် အမျိုးအစားအပေါ် သက်ရောက်မှုရှိသည်။
ကျပန်း primer method သည် နည်းလမ်းသုံးမျိုးအနက် အတိအကျအနည်းဆုံးဖြစ်သည်။တိုတောင်းသော တစ်စိတ်တစ်ပိုင်း အရှည် cDNA ထုတ်လုပ်ရန် စာသားမှတ်တမ်းတစ်လျှောက် ဆိုဒ်အများအပြားတွင် Primer များကို စုပ်ယူထားသည်။အလယ်တန်းဖွဲ့စည်းပုံဆိုင်ရာဒေသများပါရှိသော RNA နမူနာများမှ 5′ terminal sequences နှင့် cDNA ကိုရယူရန် သို့မဟုတ် reverse transcriptase ထပ်ပွားမရနိုင်သော terminating sites များနှင့် မကြာခဏအသုံးပြုပါသည်။အရှည်ဆုံး cDNA ကိုရရှိရန်၊ RNA နမူနာတစ်ခုစီရှိ primers နှင့် RNA အချိုးကို မျက်မြင်ကိုယ်တွေ့ ဆုံးဖြတ်ရန် လိုအပ်သည်။ကျပန်း primers များ၏ ကနဦးအာရုံစူးစိုက်မှုသည် 20μl တုံ့ပြန်မှုစနစ်တစ်ခုလျှင် 50 မှ 250ng မှ ကွာသည်။ကျပန်း primers များကို အဓိကအားဖြင့် ribosomal RNA ကို အသုံးပြု၍ စုစုပေါင်း RNA မှ ပေါင်းစပ်ပေါင်းစပ်ထားသော cDNA ဖြစ်သောကြောင့် poly(A)+RNA ကို ယေဘုယျအားဖြင့် ပုံစံပလိတ်အဖြစ် ရွေးချယ်ထားသည်။
Oligo(dT) အစပျိုးမှုသည် ကျပန်း primer များထက် ပိုမိုတိကျပါသည်။၎င်းသည် eukaryotic ဆဲလ်အများစုရှိ mRNA ၏ 3′ အဆုံးတွင်တွေ့ရသော poly(A) အမြီးနှင့် ပေါင်းစပ်ထားသည်။poly(A)+RNA သည် စုစုပေါင်း RNA ၏ အကြမ်းဖျင်း 1% မှ 2% ဖြစ်သောကြောင့်၊ cDNA ၏ ပမာဏနှင့် ရှုပ်ထွေးမှုသည် ကျပန်း primer ကိုအသုံးပြုပါက ပမာဏထက် များစွာနည်းပါသည်။၎င်း၏မြင့်မားသောတိကျမှုများကြောင့် oligo(dT) သည် ယေဘုယျအားဖြင့် RNA မှ primer အချိုးနှင့် poly(A)+ ရွေးချယ်မှုအတွက် ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ရန်မလိုအပ်ပါ။20μl တုံ့ပြန်မှုစနစ်အတွက် 0.5μg oligo(dT) ကို အသုံးပြုရန် အကြံပြုထားသည်။oligo(dT)12-18 သည် RT-PCR အများစုအတွက် သင့်လျော်သည်။ThermoScript RT-PCR စနစ်သည် ၎င်း၏ကောင်းမွန်သောအပူတည်ငြိမ်မှုကြောင့် oligo(dT)20 ကိုထောက်ပံ့ပေးပြီး ပိုမိုမြင့်မားသောအပူချိန်များအတွက်သင့်လျော်သည်။
Gene-specific primers (GSP) များသည် ပြောင်းပြန် ကူးယူခြင်း အဆင့်အတွက် အကောင်းဆုံး သီးသန့် primers များဖြစ်သည်။GSP သည် ကျပန်း primers သို့မဟုတ် oligo(dT) ကဲ့သို့ Rnas အားလုံးကို ဖယ်ထုတ်မည့်အစား RNA ဦးတည်ရာအစီအစဥ်များနှင့် သီးသန့်ပေါင်းစပ်နိုင်သော antisense oligonucleoside တစ်ခုဖြစ်သည်။PCR primers ဒီဇိုင်းရေးဆွဲရာတွင်အသုံးပြုသည့်စည်းမျဉ်းများသည် reverse transcription reaction GSP ၏ဒီဇိုင်းနှင့်လည်းသက်ဆိုင်ပါသည်။GSP သည် mRNA3′ ၏အဆုံးတွင် ထည့်သွင်းထားသော amplification primer နှင့် တူညီသော sequence ဖြစ်နိုင်သည်၊ သို့မဟုတ် GSP သည် reverse amplification primer ဖြင့် ရေအောက်ပိုင်းကို annealed လုပ်ရန် GSP ကို ဒီဇိုင်းထုတ်နိုင်သည်။အချို့သော ချဲ့ထွင်ထားသော အရာဝတ္ထုများအတွက်၊ အောင်မြင်သော RT-PCR အတွက် antisense primer တစ်ခုထက်ပို၍ ဒီဇိုင်းဆွဲရန် လိုအပ်သောကြောင့် ပစ်မှတ် RNA ၏ ဒုတိယဖွဲ့စည်းပုံသည် primer ကို ချိတ်တွယ်ခြင်းမှ တားဆီးနိုင်သောကြောင့် ဖြစ်သည်။20μl၏ပထမကွင်းဆက်ပေါင်းစပ်တုံ့ပြန်မှုစနစ်တွင် 1pmol antisense GSP ကိုအသုံးပြုရန်အကြံပြုထားသည်။
2. ပြောင်းပြန်ကူးယူခြင်း၏ အပူထိန်းသိမ်းမှုအပူချိန်ကို တိုးမြှင့်ပါ
GSP တိကျမှု၏ အပြည့်အဝအားသာချက်ကို ရယူရန်အတွက် မြင့်မားသောအပူတည်ငြိမ်မှုရှိသော ပြောင်းပြန်စာသားမှတ်တမ်းကို အသုံးပြုသင့်သည်။တုံ့ပြန်မှုအားကောင်းစေရန် အပူတည်ငြိမ်သောပြောင်းပြန် transcriptase ကို ပိုမိုမြင့်မားသောအပူချိန်တွင် လျှပ်ကာထားနိုင်သည်။ဥပမာအားဖြင့်၊ GSP တစ်ခုအား 55°C တွင် ဖျက်ထားလျှင် AMV သို့မဟုတ် M-MLV ကို အသုံးပြု၍ 37°C တွင် ပြောင်းပြန်ကူးယူခြင်းကို လုပ်ဆောင်ပါက GSP ၏ တိကျသေချာမှုကို အပြည့်အဝ အသုံးမချနိုင်ပါ။သို့သော်လည်း၊ SuperScripⅡ နှင့် ThermoScript သည် 50 ℃ သို့မဟုတ် ထို့ထက် ပိုသောနေရာတွင် တုံ့ပြန်နိုင်ပြီး အပူချိန်နိမ့်သောနေရာတွင် ထုတ်လုပ်သော သီးခြားမဟုတ်သော ထုတ်ကုန်များကို ဖယ်ရှားပေးသည်။အမြင့်ဆုံးတိကျမှုအတွက်၊ RNA/ primer အရောအနှောကို 65 ℃ denaturation အပူချိန်မှ အပူချိန် 2 x တုံ့ပြန်မှုအရောအနှော (cDNA ပေါင်းစပ်မှု၏အပူရှိန်စတင်ခြင်း) ပေါင်းထည့်ခြင်းဖြင့် ပြောင်းပြန်စာသားပြောင်းပြန်ကိုင်ထားသောအပူချိန်သို့ တိုက်ရိုက်လွှဲပြောင်းနိုင်သည်။၎င်းသည် အပူချိန်နိမ့်ချိန်တွင် မော်လီကျူးများကြား အခြေခံတွဲချိတ်ခြင်းကို တားဆီးပေးသည်။PCR တူရိယာကိုအသုံးပြုခြင်းသည် RT-PCR အတွက် လိုအပ်သော အပူချိန်ပြောင်းလဲမှုများစွာကို ရိုးရှင်းစေသည်။
3. မျိုးဗီဇ DNA ညစ်ညမ်းမှုကို လျှော့ချပါ
RT-PCR တွင် ဖြစ်နိုင်ချေရှိသော အခက်အခဲတစ်ခုမှာ RNA သည် မျိုးရိုးဗီဇ DNA ကို ညစ်ညမ်းစေသည်။Trizol Reagent ကဲ့သို့သော ပိုမိုကောင်းမွန်သော RNA ခွဲထုတ်နည်းများကို အသုံးပြုခြင်းသည် RNA ပြင်ဆင်မှုများတွင် မျိုးဗီဇ DNA ညစ်ညမ်းမှုကို လျော့နည်းစေသည်။မျိုးရိုးဗီဇ DNA မှ ထုတ်လုပ်သော ထုတ်ကုန်များကို ရှောင်ရှားရန်၊ RNA သည် ညစ်ညမ်းသော DNA ကို ပြောင်းပြန်မကူးမီ ဖယ်ရှားရန် ချဲ့ထွင်မှုအဆင့် DnasⅠ ဖြင့် ကုသနိုင်ပါသည်။နမူနာများကို 2.0mM EDTA တွင် 65 ℃ ဖြင့် DNaseⅠ အစာခြေခြင်းကို ရပ်တန့်ရန် 10 မိနစ်ကြာ သိမ်းဆည်းထားသည်။EDTA သည် မြင့်မားသောအပူချိန်တွင် ဖြစ်ပေါ်သော မဂ္ဂနီဆီယမ်အိုင်းယွန်းကို မှီခိုနေသော RNA hydrolysis ကို တားဆီးရန် မဂ္ဂနီဆီယမ်အိုင်းယွန်းများကို ချေဖျက်ပေးသည်။
ချဲ့ထွင်ထားသော cDNA ကို genome DNA ချဲ့ထွင်ခြင်း ထုတ်ကုန်မှ ခွဲထုတ်ရန်အတွက်၊ သီးခြား exon နှင့် သီးခြားခွဲထုတ်ထားသော primer များကို ဒီဇိုင်းထုတ်နိုင်သည်။cDNA မှရရှိသော PCR ထုတ်ကုန်များသည် ညစ်ညမ်းနေသော မျိုးရိုးဗီဇ DNA မှရရှိသော ထုတ်ကုန်များထက် ပိုတိုလိမ့်မည်။ပေးထားသောအပိုင်းအစသည် genomic DNA သို့မဟုတ် cDNA မှဖြစ်ကြောင်း ဆုံးဖြတ်ရန် RNA ပုံစံတစ်ခုစီတွင် ပြောင်းပြန်ကူးယူခြင်းမပြုဘဲ ထိန်းချုပ်ထားသော စမ်းသပ်မှုကိုလည်း လုပ်ဆောင်ပါသည်။ပြောင်းပြန်ကူးယူခြင်းမရှိဘဲ ရရှိသော PCR ထုတ်ကုန်များသည် ဂျီနိုမ်မှ ဆင်းသက်လာသည်။
ဆက်စပ်ထုတ်ကုန်
RT-PCR လွယ်ကူသည်။ᵀᴹငါ (ခြေတစ်လှမ်း)
- တစ်ဆင့်ချင်း ကိရိယာသည် ပြောင်းပြန် စာသားမှတ်တမ်းနှင့် PCR ကို တူညီသော ပြွန်ထဲတွင် လုပ်ဆောင်နိုင်စေပါသည်။၎င်းသည် နမူနာပုံစံ RNA၊ သီးခြား PCR primers နှင့် RNase-Free ddH တို့ကိုသာ ထည့်သွင်းရန် လိုအပ်သည်။2O.
- RNA ၏ အချိန်နှင့်တပြေးညီ ပမာဏခွဲခြမ်းစိတ်ဖြာမှုကို လျင်မြန်တိကျစွာ လုပ်ဆောင်နိုင်သည်။
- ပစ္စည်းကိရိယာသည် ထူးခြားသော Foregene ပြောင်းပြန် စာသားပြောင်းပြန်ဓါတ်ဆားနှင့် Foregene HotStar Taq DNA Polymerase နှင့် တုံ့ပြန်မှုစနစ်၏ ချဲ့ထွင်မှုထိရောက်မှုနှင့် တိကျမှုတို့ကို ထိထိရောက်ရောက် မြှင့်တင်ရန်အတွက် အသုံးပြုသည်။
- အကောင်းဆုံးတုံ့ပြန်မှုစနစ်သည် တုံ့ပြန်မှုကို ပိုမိုမြင့်မားသော ထောက်လှမ်းနိုင်စွမ်း၊ ပိုမိုအားကောင်းသော အပူတည်ငြိမ်မှုနှင့် သည်းခံနိုင်စွမ်းကို ပိုမြင့်မားစေသည်။
- ပုံစံခွက်အတွင်းရှိ gDNA ကို ၂ မိနစ်အတွင်း ဖယ်ရှားနိုင်သည့် gDNA ကို ထိရောက်စွာ ဖယ်ရှားနိုင်သည်။
- ထိရောက်သော ပြောင်းပြန် စာသားမှတ်တမ်းစနစ်၊ ပထမကြိုးမျှင် cDNA ၏ပေါင်းစပ်မှုကို အပြီးသတ်ရန် 15 မိနစ်သာ ကြာပါသည်။
-Complex templates- မြင့်မားသော GC အကြောင်းအရာများနှင့် ရှုပ်ထွေးသော ဆင့်ပွားတည်ဆောက်ပုံပါရှိသော တင်းပလိတ်များကို စွမ်းဆောင်ရည်မြင့်မားစွာဖြင့် ပြောင်းပြန်လှန်နိုင်သည်။
-High-sensitivity reverse transcription system၊ pg-level templates များသည် အရည်အသွေးမြင့် cDNA ကိုရနိုင်သည်။
- ပြောင်းပြန်ကူးယူခြင်းစနစ်သည် မြင့်မားသောအပူတည်ငြိမ်မှုရှိပြီး အကောင်းဆုံးတုံ့ပြန်မှုအပူချိန်မှာ 42 ℃ဖြစ်ပြီး၊ ၎င်းသည် 50 ℃တွင် ကောင်းမွန်သောပြောင်းပြန်စာသားမှတ်တမ်းစွမ်းဆောင်ရည်ရှိနေဆဲဖြစ်သည်။
စာတိုက်အချိန်- မတ်လ-၀၇-၂၀၂၃