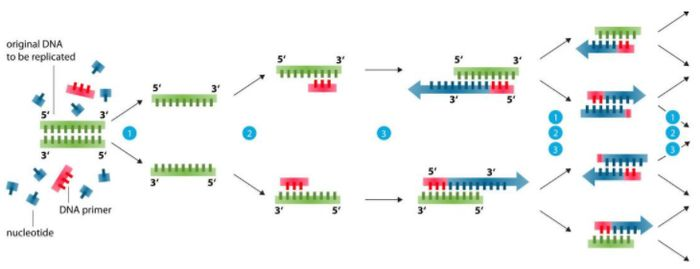
Primer ဒီဇိုင်းအခြေခံ (99% ပြဿနာများကိုဖြေရှင်းနိုင်သည်)
1. Primer length- ကျောင်းသုံးစာအုပ်သည် 15-30bp လိုအပ်သည်၊ များသောအားဖြင့် 20bp ခန့် လိုအပ်သည်။ပကတိအခြေအနေသည် တိကျသေချာစေရန် 18-24bp ဖြစ်ရန် ပိုကောင်းသည်၊ သို့သော် ပိုရှည်လေလေ၊ ပိုရှည်လေ primer သည်လည်း တိကျမှုကို လျော့ပါးစေပြီး အထွက်နှုန်းကို လျှော့ချပေးမည်ဖြစ်သည်။
2. Primer amplification span- 200-500bp သည် သင့်လျော်ပြီး အပိုင်းအစကို သီးခြားအခြေအနေများအောက်တွင် 10kb အထိ ချဲ့နိုင်သည်။
3. Primer အခြေခံ- G+C ၏ အကြောင်းအရာသည် 40-60% ဖြစ်သင့်ပြီး G+C ချဲ့ထွင်မှု အကျိုးသက်ရောက်မှု အနည်းငယ်သာ ရှိသင့်ပြီး G+C သည် အတိအကျမဟုတ်သော တီးဝိုင်းများ ပေါ်လာရန် လွယ်ကူသည်။ATGC သည် purine သို့မဟုတ် pyrimidine nucleotides 5 ခုထက်ပိုသော အစုအဝေးများကို ရှောင်ကြဉ်ခြင်းဖြင့် ကျပန်းဖြန့်ဝေခြင်းအား အကောင်းဆုံးဖြစ်သည်။တည်ငြိမ်မှုကို တိုးမြှင့်ရန် 5′ အဆုံးနှင့် အလယ်အလတ်တန်းစားများအတွက် Multi-gc သည် တည်ငြိမ်မှု၊ 3′ အဆုံးတွင် ကြွယ်ဝသော GC ကို ရှောင်ရှားရန်၊ နောက်ဆုံး အခြေစိုက်စခန်း 3 ခုအတွက် GC မရှိပါ၊ သို့မဟုတ် နောက်ဆုံး 5 အခြေစိုက်စခန်း 3 ခုအတွက် GC မရှိပါ။
4. primer များတွင် ဒုတိယဖွဲ့စည်းပုံကို ရှောင်ကြဉ်ပြီး primer နှစ်ခုကြားတွင် ဖြည့်စွက်ခြင်းကို ရှောင်ကြဉ်ပါ၊ အထူးသဖြင့် 3' အဆုံးတွင် ဖြည့်စွက်ခြင်းကို ရှောင်ကြဉ်ပါ၊ သို့မဟုတ်ပါက primer dimer ဖြစ်ပေါ်လာမည်ဖြစ်ပြီး အတိအကျမဟုတ်သော အသံချဲ့စက်များကို ထုတ်ပေးမည်ဖြစ်ပါသည်။
5. ချိတ်ဆက်မထားသော terminal bases များကြောင့် PCR ချို့ယွင်းမှုကို ရှောင်ရှားရန်၊ အထူးသဖြင့် နောက်ဆုံးနှင့် နောက်ဆုံးခြေစွပ်များ၏ 3' အဆုံးရှိ အခြေများကို တင်းကြပ်စွာ တွဲထားသင့်သည်။
6. Primers များသည် သင့်လျော်သော ခွဲထွက်သည့်နေရာများနှင့် ပေါင်းထည့်နိုင်သည် သို့မဟုတ် ချဲ့ထွင်ထားသော ပစ်မှတ်အစီအစဥ်တွင် cleavage analysis သို့မဟုတ် molecular cloning အတွက် အလွန်အကျိုးရှိသော သင့်လျော်သော cleavage sites များ ရှိသင့်ပါသည်။
7. primers များ၏ တိကျမှု- primers များသည် nucleic acid sequence database ရှိ အခြားသော sequence များနှင့် ထင်ရှားသော တူညီမှု မရှိသင့်ပါ။
8. ဆော့ဖ်ဝဲကို အသုံးပြုရန် လေ့လာပါ- PP5၊ Oligo6၊ DNAstar၊ Vector NTI၊ primer3 (ဤအွန်လိုင်းဒီဇိုင်းသည် အကောင်းဆုံးဖြစ်သည်)။
အထက်ဖော်ပြပါ အကြောင်းအရာများသည် primer ဒီဇိုင်းပြဿနာများ၏ အနည်းဆုံး 99% ကို ဖြေရှင်းပေးနိုင်ပါသည်။
primer ဒီဇိုင်း အသေးစိတ်ကို ထိန်းချုပ်ပါ။
1. Primer အရှည်
အထွေထွေ primer အရှည်သည် 18 ~ 30 bases ဖြစ်သည်။ယေဘူယျအားဖြင့် primer ၏ annealing temperature ကိုဆုံးဖြတ်ရန်အရေးကြီးဆုံးအချက်မှာ primer ၏အရှည်ဖြစ်သည်။primer ၏ လောင်စာအပူချိန်ကို ယေဘူယျအားဖြင့် ရွေးချယ်သည် (Tm တန်ဖိုး -5 ℃)၊ အချို့သည် Tm တန်ဖိုးကို တိုက်ရိုက်အသုံးပြုသည်။အောက်ဖော်ပြပါဖော်မြူလာများကို primers များ၏ annealing အပူချိန်ကိုအကြမ်းဖျင်းတွက်ချက်ရန်အသုံးပြုနိုင်ပါသည်။
primer ၏အရှည်သည် 20bp ထက်နည်းသောအခါ- [4(G+C)+2(A+T)]-5℃
primer ၏အရှည်သည် 20bp ထက်ကြီးသောအခါ: 62.3℃+0.41℃(%GC)-500/length-5℃
ထို့အပြင်၊ ဆော့ဖ်ဝဲလ်များစွာသည် annealing အပူချိန်ကိုတွက်ချက်ရန်လည်းအသုံးပြုနိုင်သည်၊ တွက်ချက်မှုနိယာမသည်ကွဲပြားလိမ့်မည်၊ ထို့ကြောင့်တခါတရံတွက်ချက်တန်ဖိုးသည်အနည်းငယ်ကွာဟနိုင်သည်။PCR တုံ့ပြန်မှုကို ပိုကောင်းအောင်ပြုလုပ်ရန်၊ 54 ℃ထက်မနည်းသော အပူချိန်ကို သေချာစေသော အတိုဆုံး primer များကို အကောင်းဆုံးထိရောက်မှုနှင့် တိကျမှုအတွက် အသုံးပြုပါသည်။
ယေဘုယျအားဖြင့်၊ primer ၏ တိကျမှုမှာ အပို nucleotide တစ်ခုစီအတွက် အချက်လေးချက်ဖြင့် တိုးလာသည်၊ ထို့ကြောင့် application အများစုအတွက် အနည်းဆုံး primer length သည် 18 nucleotides ဖြစ်သည်။primer အရှည်၏ အပေါ်ပိုင်းကန့်သတ်ချက်သည် အလွန်အရေးမကြီးပါ၊ အဓိကအားဖြင့် တုံ့ပြန်မှုထိရောက်မှုနှင့် သက်ဆိုင်ပါသည်။entropy ကြောင့်၊ primer ပိုရှည်လေ၊ DNA polymerase အတွက် တည်ငြိမ်သော double-stranded template ဖြင့် ချည်နှောင်ရန် ပစ်မှတ် DNA နှင့် ချိတ်ဆက်ရန် ၎င်းသည် တွယ်ကပ်သည့်နှုန်း နည်းပါးလေဖြစ်သည်။
primer များကိုဒီဇိုင်းရေးဆွဲရန် ဆော့ဖ်ဝဲလ်ကိုအသုံးပြုသည့်အခါ၊ primers ၏အရှည်ကို TM တန်ဖိုးဖြင့်သတ်မှတ်နိုင်ပြီး အထူးသဖြင့် fluorescence quantitative PCR၊ TM = 60 ℃ သို့မဟုတ် ထို့ထက်ပို၍ ထိန်းချုပ်သင့်သည်။
2.GC အကြောင်းအရာ
ယေဘုယျအားဖြင့်၊ primer အတွဲများတွင် G+C ၏ အကြောင်းအရာသည် 40% ~ 60% ဖြစ်ပြီး၊ primer တစ်စုံ၏ GC ပါဝင်မှုနှင့် Tm တန်ဖိုးကို ညှိနှိုင်းသင့်သည်။primer တွင် ပြင်းထန်သော GC သို့မဟုတ် AT သဘောထားရှိပါက၊ သင့်လျော်သောပမာဏ A၊ T သို့မဟုတ် G နှင့် C အမြီးကို primer ၏ 5' အဆုံးတွင် ထည့်နိုင်သည်။
3. Annealing အပူချိန်
unchain temperature ထက် 5 ℃ နိမ့်သင့်သည်။primer bases အရေအတွက် နည်းပါးပါက၊ PCR ၏ တိကျမှုကို တိုးမြင့်စေသည့် annealing temperature ကို သင့်လျော်စွာ တိုးမြှင့်နိုင်သည်။ဘေ့စ်အရေအတွက်များပါက၊ annealing temperature ကို သင့်လျော်သလို လျှော့ချနိုင်သည်။4 ℃ ~ 6 ℃ primers တစ်စုံကြားရှိအပူချိန်ခြားနားချက်သည် PCR အထွက်နှုန်းကိုမထိခိုက်စေပါ၊ သို့သော်အကောင်းဆုံးအားဖြင့် primers တစ်စုံ၏အပူပေးသည့်အပူချိန်သည်တူညီသည်၊ 55 ℃ ~ 75 ℃အကြားကွဲပြားနိုင်သည်။
4. ချဲ့ထွင်မှု ပုံစံပလိတ်၏ ဒုတိယဖွဲ့စည်းပုံ ဧရိယာကို ရှောင်ကြဉ်ပါ။
ချဲ့ထွင်ထားသောအပိုင်းကိုရွေးချယ်သောအခါ template ၏ဒုတိယဖွဲ့စည်းပုံဧရိယာကိုရှောင်ရှားရန်အကောင်းဆုံးဖြစ်သည်။ပုံစံပလိတ်ရွေးချယ်မှုအတွက် အထောက်အကူဖြစ်စေသည့် သက်ဆိုင်ရာကွန်ပျူတာဆော့ဖ်ဝဲလ်မှ ပစ်မှတ်အပိုင်းအစ၏ တည်ငြိမ်သောအလယ်တန်းဖွဲ့စည်းပုံအား ခန့်မှန်းပြီး ခန့်မှန်းနိုင်ပါသည်။တိုးချဲ့မည့်ဒေသ၏ အခမဲ့စွမ်းအင် (△G) သည် 58.6lkJ/mol ထက်နည်းသောအခါ ချဲ့ထွင်မှု မအောင်မြင်ကြောင်း စမ်းသပ်မှုရလဒ်များက ပြသသည်။
5. ပစ်မှတ် DNA နှင့် မကိုက်ညီပါ။
ချဲ့ထွင်ထားသော ပစ်မှတ် DNA အစီအစဥ်သည် ကြီးမားသောအခါ၊ primer သည် ပစ်မှတ် DNA ၏ အစိတ်အပိုင်းများစွာနှင့် ချိတ်ဆက်နိုင်ပြီး ရလဒ်တွင် ကြိုးဝိုင်းများစွာ ပေါ်လာသည်။ဤတစ်ကြိမ်တွင် BLAST ဆော့ဖ်ဝဲစမ်းသပ်ခြင်း၊ ဝဘ်ဆိုက်ကို အသုံးပြုရန် လိုအပ်သည်-http://www.ncbi.nlm.nih.gov/BLAST/.စီတန်းနှစ်ခုကို ညှိပါ (bl2seq) ကို ရွေးပါ။
ဇုန် 1 သို့ primer အစီအမံများကို ကူးထည့်ခြင်းနှင့် ဇုန် 2 သို့ ပစ်မှတ်ထားသော DNA အတွဲများကို လဲလှယ်၍ရနိုင်ပြီး BLAST သည် ဖြည့်စွက်၊ ဆန့်ကျင်ဘက်များနှင့် အခြားဖြစ်နိုင်ခြေများကို တွက်ချက်ပေးသောကြောင့် အသုံးပြုသူများသည် ကွင်းဆက်နှစ်ခုစလုံးသည် အာရုံခံကွင်းဆက်များဟုတ်မဟုတ်ကို သတိပြုမိရန် မလိုအပ်ပါ။ဒေတာဘေ့စ်ရှိ sequence ၏ GI နံပါတ်ကို သိပါက GI နံပါတ်ကိုလည်း ထည့်သွင်းနိုင်သည်၊ ထို့ကြောင့် sequence ၏ အပိုင်းကြီးတစ်ခုကို paste လုပ်ရန် မလိုအပ်ပါ။နောက်ဆုံးတွင်၊ Primer သည် ပစ်မှတ် DNA တွင် တူညီသောဆိုဒ်များစွာရှိမရှိကြည့်ရှုရန် 3 ရှိ Align ကိုနှိပ်ပါ။
6. Primer terminal
primer ၏ 3 'အဆုံးသည် extension ကိုစတင်သည့်နေရာဖြစ်သည်၊ ထို့ကြောင့်၎င်းတွင်မကိုက်ညီမှုများမှစတင်ရန်အရေးကြီးသည်။3 'အဆုံးသည် ဆက်တိုက် G သို့မဟုတ် C ထက် 3 ကြိမ်ထက် မပိုသင့်ပါ၊ အကြောင်းမှာ၊ ၎င်းသည် primer ကို G+C ဖြည့်တင်းမှုအပိုင်းတွင် မှားယွင်းစွာ အစပျိုးစေသောကြောင့် ဖြစ်သည်။အထူး PCR (AS-PCR) တုံ့ပြန်မှုများမှလွဲ၍ 3' end သည် မည်သည့်အလယ်တန်းဖွဲ့စည်းပုံမှ မဖွဲ့စည်းနိုင်ပါ၊ primer ၏ 3′ အဆုံးသည် မကိုက်ညီနိုင်ပါ။ဥပမာအားဖြင့်၊ encoding ဧရိယာကို ချဲ့ထားလျှင်၊ ချဲ့ထွင်မှု၏ တိကျမှုနှင့် ထိရောက်မှုကို ထိခိုက်စေမည့် codon ၏ တတိယအနေအထားတွင် 3 ' end ကို ရပ်တန့်သင့်ပါသည်။ပေါင်းစည်းထားသော primers ကိုအသုံးပြုသောအခါ၊ codon အသုံးပြုမှုဇယားကိုရည်ညွှန်းပါ၊ ဇီဝဗေဒဆိုင်ရာဦးစားပေးကိုအာရုံစိုက်ပါ၊ 3′ အဆုံးတွင်ထည့်သွင်းထားသော primers များကိုမသုံးပါနှင့်ပိုမိုမြင့်မားသော primers (1uM-3uM) ကိုအသုံးပြုပါ။
7. primers ၏ဒုတိယဖွဲ့စည်းပုံ
primers များသည် ဖြည့်စွက်အစီအစဥ်များ မပါဝင်သင့်ပါ၊ သို့မဟုတ်ပါက primers များသည် hairpin တည်ဆောက်ပုံများအဖြစ်သို့ ခေါက်သွားမည်ဖြစ်ပြီး၊ ဤနောက်ထပ်ဖွဲ့စည်းပုံသည် steric အတားအဆီးကြောင့် primers နှင့် templates များ၏ စည်းနှောင်မှုကို ထိခိုက်စေမည်ဖြစ်ပါသည်။အတုအယောင်စီရင်ခြင်းကို အသုံးပြုပါက၊ primers များ၏ စဉ်ဆက်မပြတ် ဖြည့်စွက်အခြေများကို ၎င်းတို့ကိုယ်တိုင် 3bp ထက် မပိုသင့်ပါ။primer နှစ်ခုကြားတွင် ပေါင်းစပ်ပါဝင်မှု မရှိသင့်ပါ၊ အထူးသဖြင့် primer dimers များဖွဲ့စည်းခြင်းကို တားဆီးရန် 3' end ၏ ဖြည့်စွက်အထပ်များကို ရှောင်ရှားသင့်သည်။ယေဘူယျအားဖြင့်၊ primers တစ်စုံကြားတွင် ဆက်တိုက်အခြေခံတူညီမှု သို့မဟုတ် ဖြည့်စွက်မှု 4 ခုထက်မပိုသင့်ပါ။
8. အမှတ်အသားများ သို့မဟုတ် loci ထည့်ပါ။
5' end သည် အသံချဲ့စက်၏ တိကျမှုအပေါ် အနည်းငယ်သက်ရောက်မှုရှိပြီး ချဲ့ထွင်မှုဆိုင်ရာ တိကျမှုကို မထိခိုက်စေဘဲ ပြုပြင်နိုင်သည်။primer 5' end ၏မွမ်းမံမှုတွင်ပါဝင်သည်- အင်ဇိုင်းကန့်သတ် site ကိုထည့်သွင်း;တံဆိပ်တပ်ထားသော biotin၊ fluorescence၊ digoxin၊ Eu3+ စသည်တို့။ ပရိုတင်းချိတ်တွယ်ထားသော DNA sequence များကို မိတ်ဆက်ပါ။ဗီဇပြောင်းလဲသည့်နေရာများကို မိတ်ဆက်ခြင်း၊ ဗီဇပြောင်းလဲမှုအစီအစဥ်များ ထည့်သွင်းခြင်းနှင့် လွဲမှားနေသော ဗီဇအစီအစဥ်များကို ထည့်သွင်းခြင်းနှင့် မြှင့်တင်ခြင်းအပိုင်းများကို မိတ်ဆက်ခြင်း စသည်ဖြင့် အပိုအခြေခံများသည် ချဲ့ထွင်ခြင်း၏ထိရောက်မှုကို အနည်းနှင့်အများအကျိုးသက်ရောက်စေမည်ဖြစ်ပြီး primer dimer ဖွဲ့စည်းနိုင်ခြေကို တိုးစေမည်ဖြစ်ပြီး၊ သို့သော် နောက်တစ်ဆင့်အတွက် လိုက်လျောမှုအချို့ပြုလုပ်ရပါမည်။ကန့်သတ်ဆိုက်များနှင့် ပရိုမိုးရှင်းအစီအစဉ်များကဲ့သို့သော ပစ်မှတ်အစီအစဥ်တွင် မရှိသော ထပ်လောင်း sequence များကို အတိအကျမထိခိုက်စေဘဲ primer ၏ 5′ အဆုံးသို့ ထည့်နိုင်သည်။ဤ sequences များသည် primer Tm တန်ဖိုးများကို တွက်ချက်ရာတွင် မပါဝင်သော်လည်း ပေါင်းစပ်ပါဝင်မှုနှင့် အတွင်းပိုင်းဒုတိယဖွဲ့စည်းပုံအတွက် စမ်းသပ်သင့်ပါသည်။
9. Subclones
အချိန်အများစုတွင် PCR သည် ပဏာမပုံတူပွားခြင်းသာဖြစ်ပြီး၊ ထို့နောက် ကျွန်ုပ်တို့သည် ပစ်မှတ်အပိုင်းအစကို အမျိုးမျိုးသော vector များအဖြစ် ခွဲထုတ်ရန် လိုအပ်သည်၊ ထို့ကြောင့် PCR အဆင့်တွင် နောက်ထပ်လုပ်ဆောင်မှုများအတွက် ထပ်လောင်းအခြေခံများကို ဒီဇိုင်းထုတ်ရန် လိုအပ်ပါသည်။
subcloning အတွက် ဒီဇိုင်းထုတ်ထားသော အချို့သော sequence များကို အောက်တွင် အကျဉ်းချုံးထားသည်။
Restriction endonuclease restriction site ကို ထည့်သွင်းခဲ့သည်။
အင်ဇိုင်းကန့်သတ်ဆိုဒ်များထည့်ခြင်းသည် PCR ထုတ်ကုန်များခွဲခြင်းအတွက် အသုံးအများဆုံးနည်းလမ်းဖြစ်သည်။ယေဘုယျအားဖြင့် cleavage site သည် six bases ဖြစ်ပြီး 5 ' cleavage site ၏အဆုံးအပြင် 2 ~ 3 protective bases ကိုပါ ထည့်သွင်းရန်လိုအပ်ပါသည်။သို့သော် မတူညီသော အင်ဇိုင်းများအတွက် လိုအပ်သော အကာအကွယ်အခြေခံ အရေအတွက် ကွဲပြားသည်။ဥပမာအားဖြင့်၊ SalⅠ အကာအကွယ်အခြေခံမလိုအပ်ပါ၊ EcoRⅤ အကာအကွယ်အခြေခံ 1 ခု လိုအပ်သည်၊ NotⅠ အကာအကွယ်အခြေခံ 2 ခု လိုအပ်ပြီး Hind Ⅲ အကာအကွယ်အခြေခံ 3 ခု လိုအပ်သည်။
LIC သည် အမြီးကို ထည့်သည်။
LIC ၏ နာမည်အပြည့်အစုံမှာ PET vector ၏ အစိတ်အပိုင်းအတွက် အထူးပြု Navogen မှ တီထွင်ခဲ့သော Ligation-Independent cloning နည်းလမ်းဖြစ်သည်။LIC နည်းလမ်းဖြင့် ပြင်ဆင်ထားသော pET သယ်ဆောင်သူတွင် ဖြည့်စွက်မဟုတ်သော 12-15 အခြေခံတစ်ခုတည်းသော ကပ်စေးကပ်သောစွန်းများပါရှိပြီး ပစ်မှတ်ထည့်သွင်းသည့်အပိုင်းတွင် သက်ဆိုင်ရာ ကပ်စေးနေသောစွန်းများကို ဖြည့်စွက်ပေးသည်။ချဲ့ထွင်ရန် ရည်ရွယ်ချက်များအတွက်၊ ထည့်သွင်းထားသောအပိုင်းအစ၏ primer 5′ အစီစဥ်သည် LIC vector ကို ဖြည့်စွက်သင့်သည်။T4 DNA polymerase ၏ 3′→5′ ထုတ်ယူထားသော လုပ်ဆောင်ချက်သည် အချိန်တိုအတွင်း ထည့်သွင်းထားသော အပိုင်းအစတွင် ကပ်စေးကပ်သော ကြိုးမျှင်တစ်ခုတည်းကို ဖန်တီးနိုင်သည်။ထုတ်ကုန်သည် ပြင်ဆင်ထားသော အပိုင်းအစများနှင့် vector ကို အပြန်အလှန် ပေါင်းထည့်ခြင်းဖြင့်သာ ဖွဲ့စည်းနိုင်သောကြောင့်၊ ဤနည်းလမ်းသည် အလွန်မြန်ဆန်ပြီး ထိရောက်ပြီး ၎င်းကို ပုံတူကူးရန် ညွှန်ကြားထားသည်။
TA အမြီးထည့်ရန် ညွှန်ကြားထားသည်။
TA cloning သည် အပိုင်းတစ်ပိုင်းကို vector တစ်ခုအဖြစ် ပစ်မှတ်ထားနိုင်ခြင်း မရှိခဲ့သဖြင့် နောက်ပိုင်းတွင် Invitrogen သည် တစ်ဖက်တွင် ထင်ရှားသော အခြေခံ GTGGS လေးခုပါရှိသော ပုံတူပွားခြင်းကို ပစ်မှတ်ထားနိုင်သည့် vector တစ်ခုကို မိတ်ဆက်ပေးခဲ့သည်။ထို့ကြောင့် PCR primers များ၏ဒီဇိုင်းတွင်၊ အပိုင်းအစများကို "ဦးတည်" နိုင်စေရန်အတွက် ဖြည့်စွက်အစီအမံများကို အလိုက်သင့်ထည့်သွင်းသင့်သည်။
အချိန်တိုအတွင်း ဗီဇကို vector နှင့် ပေါင်းစပ်ပြီး ကြွက်သားပညာရှင်များတွင် ET gene synthesis ဟုခေါ်သော တိုက်ရိုက်ပေါင်းစပ်မှုကို စမ်းကြည့်နိုင်ပါသည်။
D. In-Fusion ပုံတူပွားနည်း
ligase မလိုအပ်ပါ၊ ကြာရှည်စွာတုံ့ပြန်မှုမလိုအပ်ပါ။linearized vector ၏အစွန်းနှစ်ဖက်ရှိ sequence ကို primers ၏ဒီဇိုင်းတွင်မိတ်ဆက်ထားသရွေ့ PCR ထုတ်ကုန်နှင့် linearized vector ကို BSA ပါ ၀ င်သော in-fusion အင်ဇိုင်းဖြေရှင်းချက်ထဲသို့ထည့်သွင်းပြီးနာရီဝက်ကြာအခန်းအပူချိန်တွင်ထားရှိခြင်းဖြင့်အသွင်ပြောင်းမှုကိုလုပ်ဆောင်နိုင်သည်။ဤနည်းလမ်းသည် အသံအတိုးအကျယ်ပြောင်းလဲခြင်းအတွက် အထူးသင့်လျော်ပါသည်။
10. primer ပေါင်းလိုက်ပါ။
တစ်ခါတစ်ရံတွင် primer ဒီဇိုင်းအကြောင်း အကန့်အသတ်ရှိသော sequence အချက်အလက်ကိုသာ သိသည်။ဥပမာအားဖြင့်၊ အမိုင်နိုအက်ဆစ် sequence ကိုသာသိပါက၊ ပေါင်းစပ် primer ကိုဒီဇိုင်းထုတ်နိုင်သည်။ပေါင်းစပ် primer သည် တစ်ခုတည်းသော အမိုင်နိုအက်ဆစ်ကို ကုဒ်လုပ်သည့် မတူညီသော အခြေခံဖြစ်နိုင်ခြေအားလုံးကို ကိုယ်စားပြုသည့် ကွဲပြားသော sequence များ၏ ရောနှောမှုတစ်ခုဖြစ်သည်။တိကျသေချာစေရန်၊ မတူညီသောသက်ရှိများ၏ အခြေခံအသုံးပြုမှုဦးစားပေးများအလိုက် ပေါင်းစပ်ထည့်သွင်းမှုကို လျှော့ချရန်အတွက် codon အသုံးပြုမှုဇယားကို ကိုးကားနိုင်ပါသည်။primer ၏ annealing အပူချိန်ကိုလျှော့ချရန်အတွက် Hypoxanthine ကို bases အားလုံးနှင့်တွဲနိုင်သည်။Primer ၏ 3′ အဆုံးတွင် ချိတ်ထားသော base များကို 3′ အဆုံးတွင် ပေါင်းထည့်ခြင်းသည် မှားယွင်းသော site တွင် PCR စတင်ရန် လုံလောက်သောကြောင့် မသုံးပါနှင့်။ပေါင်းစည်းထားသော အရောအနှောများစွာရှိ primer များသည် ပစ်မှတ်ပုံစံပလိတ်အတွက် သီးခြားမဟုတ်သောကြောင့် ပိုမိုမြင့်မားသော primer ပြင်းအား (1μM မှ 3μM) ကို အသုံးပြုပါသည်။
PCR ကုန်ကြမ်းထိန်းချုပ်မှု
1. Primer ပမာဏ
primer တစ်ခုစီ၏အာရုံစူးစိုက်မှုသည် 0.1 ~ 1umol သို့မဟုတ် 10 ~ 100pmol ဖြစ်သည်။Primer အနိမ့်ဆုံးနဲ့ လိုအပ်တဲ့ ရလဒ်ကို ထုတ်ပေးတာက ပိုကောင်းပါတယ်။Primer ၏ ပြင်းအား မြင့်မားခြင်းသည် ကိုက်ညီမှုမရှိသော ချဲ့ထွင်မှုကို ဖြစ်စေပြီး primer များကြားတွင် dimers များဖွဲ့စည်းနိုင်ခြေကို တိုးစေသည်။
2. Primer အာရုံစူးစိုက်မှု
Primers ၏အာရုံစူးစိုက်မှုသည်တိကျမှုကိုအကျိုးသက်ရောက်သည်။အကောင်းဆုံး primer အာရုံစူးစိုက်မှုသည် ယေဘူယျအားဖြင့် 0.1 နှင့် 0.5μM ကြားဖြစ်သည်။ပိုမြင့်သော primer ပါဝင်မှုသည် အတိအကျမဟုတ်သော ထုတ်ကုန်များကို ချဲ့ထွင်ရန် ဦးတည်သည်။
3. Primer ၏ အပူချိန်ကို စုပ်ယူခြင်း။
primers အတွက် နောက်ထပ်အရေးကြီးသော parameter သည် အရည်ပျော်သည့်အပူချိန် (Tm) ဖြစ်သည်။primers ၏ 50% နှင့် complementary sequences များကို double-stranded DNA မော်လီကျူးများအဖြစ် ကိုယ်စားပြုသောအခါ ဤအပူချိန်ဖြစ်သည်။PCR annealing အပူချိန်သတ်မှတ်ရန် Tm လိုအပ်သည်။အကောင်းဆုံးကတော့၊ ပစ်မှတ်အစီအစဥ်နဲ့ primers တွေကို ထိထိရောက်ရောက် လိမ်းကျံဖို့ သေချာအောင် လိမ်းပေးရတဲ့ အပူချိန်က နိမ့်နေပေမယ့် အတိအကျမဟုတ်တဲ့ ချည်နှောင်မှုကို လျှော့ချဖို့ လုံလောက်ပါတယ်။ကျိုးကြောင်းဆီလျော်သော annealing အပူချိန် 55 ℃မှ 70 ℃။Annealing temperature ကို ယေဘူယျအားဖြင့် primer ၏ Tm ထက် 5 ℃ နိမ့်သည်။
အသုံးပြုထားသောဖော်မြူလာနှင့် primers ၏အစီအစဥ်ပေါ်မူတည်၍ Tm သတ်မှတ်ခြင်းအတွက် ဖော်မြူလာများစွာရှိပါသည်။ဖော်မြူလာအများစုသည် ခန့်မှန်းခြေ Tm တန်ဖိုးကို ပေးဆောင်သောကြောင့်၊ အပူချိန်အားလုံးသည် စမှတ်တစ်ခုသာဖြစ်သည်။annealing အပူချိန်ကို အဆင့်ဆင့်မြှင့်တင်ပေးသည့် တုံ့ပြန်မှုများစွာကို ခွဲခြမ်းစိတ်ဖြာခြင်းဖြင့် တိကျမှုကို မြှင့်တင်နိုင်ပါသည်။ခန့်မှန်းခြေ Tm-5 ℃ အောက်တွင် စတင်ကာ 2 ℃ တိုးမြင့်လာကာ အပူချိန်ကို ဖြည်းဖြည်းချင်း တိုးပေးပါ။ပိုမြင့်သော အပူအအေးခံခြင်းသည် primer dimers နှင့် သီးခြားမဟုတ်သော ထုတ်ကုန်များဖွဲ့စည်းခြင်းကို လျှော့ချပေးသည်။အကောင်းဆုံးရလဒ်များအတွက်၊ primer နှစ်ခုတွင် ခန့်မှန်းခြေ Tm တန်ဖိုးများ ရှိသင့်သည်။primer အတွဲများ၏ Tm ခြားနားချက်သည် 5 ℃ ထက်ပိုပါက၊ primer များသည် စက်ဝိုင်းအတွင်း အောက်ပိုင်း annealing temperature ကိုအသုံးပြုခြင်းဖြင့် primer များသည် သိသာထင်ရှားသောမှားယွင်းသောစတင်မှုကိုပြသမည်ဖြစ်သည်။primers Tm နှစ်ခု မတူပါက၊ အနိမ့်ဆုံး Tm ထက် 5 ℃ ဖြင့် annealing temperature ကို သတ်မှတ်ပါ။တနည်းအားဖြင့် တိကျသေချာစေရန်အတွက်၊ Tm မြင့်ရန်အတွက် ဒီဇိုင်းထုတ်ထားသော လောင်စာအပူချိန်တွင် သံသရာငါးခုကို ဦးစွာလုပ်ဆောင်နိုင်ပြီး၊ ၎င်းနောက်တွင် ကျန်ရှိသော စက်ဝန်းများကို Tm အောက်အတွက် ဒီဇိုင်းထုတ်ထားသော လောင်ကျွမ်းမှုအပူချိန်တွင် လည်ပတ်နိုင်သည်။၎င်းသည် တင်းကျပ်သောအခြေအနေအောက်တွင် ဦးတည်ရာပုံစံပလိတ်၏ တစ်စိတ်တစ်ပိုင်းမိတ္တူကို ရယူခွင့်ပြုသည်။
4. Primer သန့်ရှင်းမှုနှင့် တည်ငြိမ်မှု
စိတ်ကြိုက် primers များ၏ စံသန့်စင်မှုသည် PCR အပလီကေးရှင်းအများစုအတွက် လုံလောက်ပါသည်။benzoyl နှင့် isobutylyl အုပ်စုများကို ဖယ်ထုတ်ခြင်းသည် အနည်းငယ်မျှသာဖြစ်ပြီး ထို့ကြောင့် PCR ကို အနှောင့်အယှက်မဖြစ်စေပါ။အချို့သော အပလီကေးရှင်းများသည် ပေါင်းစပ်မှုလုပ်ငန်းစဉ်တွင် အပြည့်အဝမဟုတ်သော အတွဲများကို ဖယ်ရှားရန် သန့်စင်ရန် လိုအပ်သည်။DNA ပေါင်းစပ်မှု ဓာတုဗေဒ၏ ထိရောက်မှု 100% မဟုတ်သောကြောင့် ဖြတ်တောက်ထားသော ဤအစီအစဥ်များသည် ဖြစ်ပေါ်လာသည်။၎င်းသည် DNA မှ 3′ မှ 5′ ထိဖြစ်အောင် base တစ်ခုစီကို ပေါင်းထည့်ထားသောကြောင့် ထပ်ခါတလဲလဲ ဓာတုတုံ့ပြန်မှုများကို အသုံးပြုသည့် စက်ဝိုင်းလုပ်ငန်းစဉ်တစ်ခုဖြစ်သည်။သံသရာမှာရော ကျရှုံးနိုင်ပါတယ်။ပိုရှည်သော primer များ၊ အထူးသဖြင့် base 50 ထက်ကြီးသော၊ ဖြတ်တောက်ထားသော sequences အများအပြားရှိပြီး သန့်စင်ရန် လိုအပ်နိုင်ပါသည်။
ဓာတုဓာတုဗေဒနှင့် သန့်စင်မှုနည်းလမ်းများ၏ ထိရောက်မှုဖြင့် primers များ၏ အထွက်နှုန်းကို ထိခိုက်ပါသည်။Cytology နှင့် Shengong ကဲ့သို့သော ဇီဝဆေးဝါးကုမ္ပဏီများသည် oligonucleoside ၏ စုစုပေါင်းထွက်ရှိမှုကို သေချာစေရန် အနိမ့်ဆုံး OD ယူနစ်ကို အသုံးပြုကြသည်။စိတ်ကြိုက် primer များကို အခြောက်မှုန့်ပုံစံဖြင့် တင်ပို့ပါသည်။နောက်ဆုံး အာရုံစူးစိုက်မှုသည် 100μM ဖြစ်စေရန် TE တွင် primers များကို ပြန်လည်ပျော်ဝင်ရန် အကောင်းဆုံးဖြစ်သည်။ရေ၏ pH သည် မကြာခဏ အက်စစ်ဓာတ်ရှိပြီး oligonucleosides ၏ hydrolysis ကို ဖြစ်စေသောကြောင့် TE သည် deionized water ထက် ပိုကောင်းပါသည်။
primers များ၏တည်ငြိမ်မှုသည်သိုလှောင်မှုအခြေအနေများပေါ်တွင်မူတည်သည်။အမှုန့်ခြောက်နှင့် အရည်ဖျော် primers များကို -20 ℃ တွင် သိမ်းဆည်းထားသင့်သည်။10μM ထက်ကြီးသောပြင်းအား TE တွင်ပျော်ဝင်နေသော primers များကို -20 ℃ 6 လကြာတည်ငြိမ်စွာသိမ်းဆည်းနိုင်သော်လည်း 1 ပတ်ထက်နည်းသောအခန်းအပူချိန် (15 ℃မှ 30 ℃) တွင်သာသိမ်းဆည်းနိုင်သည်။ခြောက်သွေ့သောအမှုန့် primers သည် -20 C တွင်အနည်းဆုံး 1 နှစ်နှင့်အခန်းအပူချိန် (15 C မှ 30 C) တွင် 2 လအထိသိမ်းဆည်းနိုင်သည်။
5. အင်ဇိုင်းများနှင့်၎င်းတို့၏ပါဝင်မှု
လက်ရှိတွင်၊ အသုံးပြုထားသော Taq DNA polymerase သည် အခြေခံအားဖြင့် coliform ဘက်တီးရီးယားများဖြင့် ပေါင်းစပ်ထုတ်လုပ်ထားသော gene engineering enzyme ဖြစ်သည်။ပုံမှန် PCR တုံ့ပြန်မှုကို ဓာတ်ကူပြုရန်အတွက် လိုအပ်သော အင်ဇိုင်းပမာဏသည် 2.5U ခန့် (စုစုပေါင်း 100ul ၏ တုံ့ပြန်မှုပမာဏကို ရည်ညွှန်းသည်)။အာရုံစူးစိုက်မှု အလွန်မြင့်မားပါက၊ အတိအကျမဟုတ်သော ချဲ့ထွင်မှုကို ဖြစ်ပေါ်စေနိုင်သည်။အာရုံစူးစိုက်မှု အလွန်နည်းပါက၊ ဓာတုပစ္စည်း ပမာဏ လျော့ကျသွားမည်ဖြစ်သည်။
6. dNTP ၏ အရည်အသွေးနှင့် အာရုံစူးစိုက်မှု
dNTP ၏အရည်အသွေးသည် အာရုံစူးစိုက်မှုနှင့် PCR ချဲ့ထွင်မှု၏ထိရောက်မှုတို့နှင့် နီးကပ်စွာဆက်စပ်နေသည်။dNTP အမှုန့်သည် အသေးအမွှားဖြစ်ပြီး၊ ၎င်းကို မှားယွင်းစွာ သိမ်းဆည်းပါက ၎င်း၏ ဇီဝဗေဒဆိုင်ရာ လုပ်ဆောင်ချက် ပျက်ယွင်းပါသည်။dNTP ဖြေရှင်းချက်သည် အက်စစ်ဓာတ်ဖြစ်ပြီး ၎င်း၏ PH 7.0 ~ 7.5 သို့ ချိန်ညှိရန် 1M NaOH သို့မဟုတ် 1M Tris.HCL ကြားခံဖြေရှင်းချက်ဖြင့် မြင့်မားသောအာရုံစူးစိုက်မှုတွင် အသုံးပြုသင့်သည်။အမြောက်အမြား အေးခဲနေသော ရောနှောခြင်းသည် dNTP ကို ကျဆင်းစေမည်ဖြစ်သည်။PCR တုံ့ပြန်မှုတွင် dNTP သည် 50 ~ 200umol/L ဖြစ်သင့်သည်။အထူးသဖြင့်၊ DNTPS လေးခု၏အာရုံစူးစိုက်မှုသည် တူညီသင့်သည် (တူညီသောမှဲ့ပြင်ဆင်မှု) ကိုအာရုံစိုက်သင့်သည်။၎င်းတို့အနက်မှ တစ်ဦးတစ်ယောက်၏ အာရုံစူးစိုက်မှုမှာ အခြားသူများနှင့် မတူပါက (အထက် သို့မဟုတ် အောက်) မကိုက်ညီမှုများ ဖြစ်ပေါ်မည်။အလွန်နည်းသော အာရုံစူးစိုက်မှုသည် PCR ထုတ်ကုန်များ၏ အထွက်နှုန်းကို လျော့နည်းစေသည်။dNTP သည် Mg2+ နှင့် ပေါင်းစပ်နိုင်ပြီး အခမဲ့ Mg2+ ၏ အာရုံစူးစိုက်မှုကို လျှော့ချနိုင်သည်။
7. ပုံစံ (ပစ်မှတ် ဗီဇ) နူကလိယအက်ဆစ်
နမူနာပုံစံ နျူကလိစ်အက်ဆစ်၏ ပမာဏနှင့် သန့်စင်မှုဒီဂရီသည် PCR ၏အောင်မြင်မှု သို့မဟုတ် ကျရှုံးမှုအတွက် အဓိကချိတ်ဆက်မှုတစ်ခုဖြစ်သည်။သမားရိုးကျ DNA သန့်စင်မှုနည်းလမ်းများသည် နမူနာများကို ချေဖျက်ရန်နှင့် စွန့်ပစ်ရန် SDS နှင့် protease K ကို အသုံးပြုလေ့ရှိသည်။SDS ၏အဓိကလုပ်ဆောင်ချက်များမှာ- ဆဲလ်အမြှေးပါးပေါ်ရှိ lipid နှင့် ပရိုတင်းများကို ပျော်ဝင်စေပြီး အမြှေးပါးပရိုတင်းများကို ပျော်ဝင်စေခြင်းဖြင့် ဆဲလ်အမြှေးပါးကို ဖျက်ဆီးကာ ဆဲလ်အတွင်းရှိ နျူကလီးယားပရိုတိန်းများကို ခွဲထုတ်နိုင်သည်၊ SDS သည် ပရိုတင်းများနှင့်လည်း ပေါင်းစပ်နိုင်သည်၊Protease K သည် DNA နှင့် ချည်နှောင်ထားသော အထူးသဖြင့် ဟစ်စတုန်းများကို hydrolyze နှင့် ချေဖျက်နိုင်ပြီး ပရိုတင်းများနှင့် အခြားသော ဆဲလ်အစိတ်အပိုင်းများကို ထုတ်ယူရန်အတွက် အော်ဂဲနစ်ပျော်ဝင်သည့် ဖီနောနှင့် ကလိုရိုဖောင်တို့ကို အသုံးပြုကာ နျူကလိယအက်ဆစ်ကို ရွာသွန်းစေရန် အီသနော သို့မဟုတ် isopropyl အရက်ကို အသုံးပြုသည်။ထုတ်ယူထားသော nucleic acid ကို PCR တုံ့ပြန်မှုများအတွက် ပုံစံတစ်ခုအဖြစ် အသုံးပြုနိုင်ပါသည်။ယေဘူယျလက်တွေ့စမ်းသပ်မှုနမူနာများအတွက်၊ ဆဲလ်များကိုပျော်ဝင်ရန်၊ lysate ရောဂါပိုးများကို lysate၊ ခရိုမိုဆုန်းမှပရိုတင်းများကိုချေဖျက်ပြီး ပစ်မှတ်မျိုးဗီဇများအထိ အမြန်နှင့်ရိုးရှင်းသောနည်းလမ်းကိုအသုံးပြုကာ PCR ချဲ့ထွင်ရန်အတွက် တိုက်ရိုက်အသုံးပြုနိုင်သည်။RNA ပုံစံပလိတ်ထုတ်ယူခြင်းသည် အများအားဖြင့် RNase အား ကျဆင်းခြင်းမှကာကွယ်ရန် guanidine isothiocyanate သို့မဟုတ် protease K နည်းလမ်းကို အသုံးပြုသည်။
8.Mg2+ အာရုံစူးစိုက်မှု
Mg2+ သည် PCR ချဲ့ထွင်မှု၏ တိကျမှုနှင့် အထွက်နှုန်းအပေါ် သိသာထင်ရှားသော သက်ရောက်မှုရှိသည်။ယေဘုယျအားဖြင့် PCR တုံ့ပြန်မှုတွင်၊ အမျိုးမျိုးသော dNTP ၏အာရုံစူးစိုက်မှုသည် 200umol/L ဖြစ်သောအခါ၊ သင့်လျော်သော Mg2+ သည် 1.5 ~ 2.0mmol/L ဖြစ်သည်။Mg2+ အာရုံစူးစိုက်မှု မြင့်မားလွန်းသည်၊ တုံ့ပြန်မှု တိကျမှု လျော့နည်းသွားသည်၊ အတိအကျ မဟုတ်သော ချဲ့ထွင်မှု ဖြစ်ပေါ်သည်၊ အာရုံစူးစိုက်မှု နည်းပါးလွန်းပါက Taq DNA polymerase ၏ လုပ်ဆောင်ချက်ကို လျော့နည်းစေပြီး တုံ့ပြန်မှု ထုတ်ကုန်များကို လျှော့ချပေးသည်။
မဂ္ဂနီဆီယမ်အိုင်းယွန်းများသည် အထွက်နှုန်းကို ထိခိုက်စေသည့် DNA polymerase လုပ်ဆောင်ချက်ကဲ့သို့သော PCR ၏ ရှုထောင့်များစွာကို သက်ရောက်မှုရှိသည်။နောက်ဥပမာတစ်ခုကတော့ primer annealing ဟာ တိကျမှုကို သက်ရောက်မှုရှိပါတယ်။dNTP နှင့် ပုံစံခွက်သည် အင်ဇိုင်းလုပ်ဆောင်ချက်အတွက် လိုအပ်သော အခမဲ့မဂ္ဂနီဆီယမ်အိုင်းယွန်းကို မဂ္ဂနီဆီယမ်အိုင်းယွန်းနှင့် ပေါင်းစပ်ထားသည်။အကောင်းဆုံးမဂ္ဂနီဆီယမ်အိုင်းယွန်းအာရုံစူးစိုက်မှုမှာ မတူညီသော primer အတွဲများနှင့် နမူနာပုံစံများအတွက် ကွဲပြားသော်လည်း 200μM dNTP ပါရှိသော ပုံမှန် PCR သည် 1.5mM ဖြစ်သည် (မှတ်ချက်- အချိန်နှင့်တစ်ပြေးညီ ပမာဏ PCR အတွက်၊ fluorescent probe ဖြင့် 3 မှ 5mM မဂ္ဂနီဆီယမ်အိုင်းယွန်းဖြေရှင်းချက်ကို အသုံးပြုပါ)။အလကားမဂ္ဂနီဆီယမ်အိုင်းယွန်းများ မြင့်မားစွာပါဝင်မှုသည် အထွက်နှုန်းကို တိုးစေပြီး အတိအကျမဟုတ်သော ချဲ့ထွင်မှုကို တိုးမြင့်စေပြီး သစ္စာရှိမှုကို ကျဆင်းစေသည်။အကောင်းဆုံးအာရုံစူးစိုက်မှုအား ဆုံးဖြတ်ရန်၊ မဂ္ဂနီဆီယမ်အိုင်းယွန်း တိုင်ထရိတ်များကို 1mM မှ 3mM မှ 0.5mM သို့ တိုး၍လုပ်ဆောင်ခဲ့သည်။မဂ္ဂနီဆီယမ်အိုင်းယွန်း ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ခြင်းအပေါ် မှီခိုမှုကို လျှော့ချရန်အတွက် Platinum Taq DNA polymerase ကို အသုံးပြုနိုင်သည်။Platinum Taq DNA polymerase သည် Taq DNA polymerase ထက် မဂ္ဂနီဆီယမ်အိုင်းယွန်းပြင်းအား ကျယ်ပြန့်စွာလုပ်ဆောင်နိုင်သောကြောင့် ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ရန် လိုအပ်ပါသည်။
9. Pcr-မြှင့်တင်သော ဖြည့်စွက်ပစ္စည်းများ
အပူချိန်၊ primer ဒီဇိုင်းနှင့် မဂ္ဂနီဆီယမ်အိုင်းယွန်း အာရုံစူးစိုက်မှုတို့ကို အကောင်းဆုံးဖြစ်အောင် ပြုလုပ်ခြင်းသည် ပုံစံပလိတ်အများစု၏ အလွန်တိကျသော ချဲ့ထွင်မှုအတွက် လုံလောက်ပါသည်။သို့သော်လည်း၊ GC မြင့်မားသော အကြောင်းအရာများ အပါအဝင် အချို့သော နမူနာပုံစံများသည် ထပ်လောင်းတိုင်းတာမှုများ လိုအပ်ပါသည်။DNA ၏ အရည်ပျော်မှု အပူချိန်ကို ထိခိုက်စေသော ပေါင်းထည့်ပစ္စည်းများသည် ထုတ်ကုန်၏ တိကျမှုနှင့် အထွက်နှုန်းကို မြှင့်တင်ရန် အခြားနည်းလမ်းကို ပေးစွမ်းသည်။အကောင်းဆုံးရလဒ်များအတွက် နမူနာပုံစံ၏ အသွင်အပြင် အပြည့်အစုံ လိုအပ်သည်။
ထို့အပြင်၊ ဒုတိယဖွဲ့စည်းပုံသည် primer binding နှင့် enzyme extension ကိုတားဆီးသည်။
formamide၊ DMSO၊ glycerin၊ betaine နှင့် PCRx Enhancer Solution အပါအဝင် PCR ဖြည့်စွက်ပစ္စည်းများသည် ချဲ့ထွင်မှုကို မြှင့်တင်ပေးသည်။၎င်းတို့၏ ဖြစ်နိုင်ချေရှိသော ယန္တရားမှာ အရည်ပျော်မှု အပူချိန်ကို လျှော့ချရန်ဖြစ်ပြီး၊ ထို့ကြောင့် primers များကို ပေါင်းထည့်ခြင်းနှင့် အလယ်တန်းဖွဲ့စည်းပုံ ဒေသမှတဆင့် DNA polymerase extension ကို ကူညီပေးသည်။PCRx Solution တွင် အခြားအားသာချက်များရှိသည်။Platinum Taq DNA polymerase နှင့် Platinum Pfx DNA polymerase တို့နှင့် အသုံးပြုသောအခါ အနည်းဆုံး မဂ္ဂနီဆီယမ်အိုင်းယွန်း ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ရန် လိုအပ်ပါသည်။ထို့ကြောင့်၊ ပလက်တီနမ်နည်းပညာကို တတိယနည်းလမ်းဖြစ်သော မဂ္ဂနီဆီယမ်အိုင်းယွန်း ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ခြင်း၏ မှီခိုမှုကို လျှော့ချနေစဉ် တိကျမှုတိုးစေရန် ပေါင်းထည့်ခြင်းဖြင့် ပေါင်းစပ်ထားသည်။အကောင်းဆုံးရလဒ်များအတွက်၊ အထူးသဖြင့် Taq DNA polymerase ကိုတားစီးပေးသော DMSO၊ formamide နှင့် glycerol ပေါင်းထည့်ထားသော အာရုံစူးစိုက်မှုကို အကောင်းဆုံးဖြစ်အောင်ပြုလုပ်သင့်သည်။
10. ပူစတင်
Hot start PCR သည် ကောင်းမွန်သော primer ဒီဇိုင်းအပြင် PCR တိကျမှုကို မြှင့်တင်ရန် အရေးကြီးဆုံးနည်းလမ်းများထဲမှတစ်ခုဖြစ်သည်။Taq DNA polymerase ၏ အကောင်းဆုံး ရှည်လျားသော အပူချိန်မှာ 72 ℃ ဖြစ်သော်လည်း၊ ပေါ်လီမာရစ်သည် အခန်းအပူချိန်တွင် ဆက်လက် လှုပ်ရှားနေပါသည်။ထို့ကြောင့် PCR တုံ့ပြန်မှုပြင်ဆင်ချိန်နှင့် အပူစက်ဝန်းအစတွင် ကိုင်ဆောင်ထားသောအပူချိန်ထက် အပူချိန်နိမ့်သည့်အခါ အတိအကျမဟုတ်သော ထုတ်ကုန်များကို ထုတ်လုပ်သည်။ဖွဲ့စည်းပြီးသည်နှင့်၊ ဤမသတ်မှတ်ထားသောထုတ်ကုန်များကို ထိထိရောက်ရောက် ချဲ့ထွင်သည်။ဆိုဒ်မှညွှန်ကြားသော ဗီဇပြောင်းလဲခြင်း၊ ထုတ်ဖော်ပြောဆိုပုံပွားခြင်း သို့မဟုတ် တည်ဆောက်ခြင်းနှင့် DNA အင်ဂျင်နီယာအတွက် အသုံးပြုသော မျိုးရိုးဗီဇဆိုင်ရာဒြပ်စင်များကဲ့သို့ မျိုးရိုးဗီဇဒြပ်စင်များ၏တည်နေရာကို ကန့်သတ်ထားသည့်အခါ Hot-start PCR သည် အထူးထိရောက်ပါသည်။
Taq DNA polymerase ၏ လုပ်ဆောင်မှုကို ကန့်သတ်ရန် ဘုံနည်းလမ်းမှာ ရေခဲပေါ်တွင် PCR တုံ့ပြန်မှုဖြေရှင်းချက်ကို ပြင်ဆင်ပြီး ကြိုတင်အပူပေးထားသော PCR ယန္တရားတစ်ခုတွင် ထားခြင်းဖြစ်သည်။ဤနည်းလမ်းသည် ရိုးရှင်းပြီး စျေးမကြီးသော်လည်း ၎င်းသည် အင်ဇိုင်း၏လုပ်ဆောင်ချက်ကို ပြီးမြောက်အောင် မလုပ်ဆောင်နိုင်သောကြောင့် သီးသန့်မဟုတ်သော ထုတ်ကုန်များ၏ ချဲ့ထွင်မှုကို လုံးလုံးလျားလျား မဖယ်ရှားနိုင်ပါ။
PCR ယန္တရားသည် denaturation temperature မရောက်မချင်း မရှိမဖြစ်လိုအပ်သော အစိတ်အပိုင်းတစ်ခုကို ဟန့်တားခြင်းဖြင့် DNA ပေါင်းစပ်မှုကို နှောင့်နှေးစေသည်။Taq DNA polymerase ကို နှောင့်နှေးအောင် ပေါင်းထည့်ခြင်း အပါအဝင် လက်စွဲအပူ စတင်ခြင်းနည်းလမ်း အများစုသည် အထူးသဖြင့် high-throughput applications များအတွက် ခက်ခဲသည်။အခြားအပူခံနည်းလမ်းများသည် မဂ္ဂနီဆီယမ်အိုင်းယွန်း သို့မဟုတ် အင်ဇိုင်းများအပါအဝင် မရှိမဖြစ်လိုအပ်သောအစိတ်အပိုင်းကို ဖုံးအုပ်ရန် သို့မဟုတ် ပုံစံပလိတ်များနှင့် ကြားခံများကဲ့သို့သော ဓာတ်ပြုအစိတ်အပိုင်းများကို ရုပ်ပိုင်းအရခွဲထုတ်ရန် ဖယောင်းအကာကို အသုံးပြုသည်။အပူစက်ဝန်းအတွင်း ဖယောင်းအရည်ပျော်သွားသည်နှင့်အမျှ အစိတ်အပိုင်းအမျိုးမျိုးကို ထုတ်လွှတ်ကာ ရောနှောသွားကြသည်။manual hot start method ကဲ့သို့ပင် wax shield method သည် ရှုပ်ထွေးပြီး ညစ်ညမ်းနိုင်ခြေများပြီး high throughput applications များအတွက် မသင့်လျော်ပါ။
Platinum DNA polymerase သည် အလိုအလျောက် hot start PCR အတွက် အဆင်ပြေပြီး ထိရောက်သည်။Platinum Taq DNA polymerase သည် Taq DNA polymerase ဆန့်ကျင်ဘက် monoclonal antibody နှင့် ပေါင်းစပ်ထားသော Taq DNA polymerase ပါဝင်သည်။ပဋိပစ္စည်းများကို အချိန်အကြာကြီး အပူချိန်ထိန်းထားချိန်တွင် အင်ဇိုင်းလုပ်ဆောင်ချက်ကို ဟန့်တားရန် PCR မှ ဖော်စပ်ထားသည်။Taq DNA polymerase သည် denaturation အဆင့်၏ 94 ℃ insulation တွင် တုံ့ပြန်မှုထဲသို့ ထုတ်လွှတ်ပြီး အပြည့်အဝ polymerase လုပ်ဆောင်မှုကို ပြန်လည်ရရှိစေသည်။ဓာတုဗေဒနည်းအရ ပြုပြင်ထားသော Taq DNA polymerase နှင့် ဆန့်ကျင်ဘက်အနေနှင့်၊ ပလတ်တီနမ်အင်ဇိုင်းသည် ပေါ်လီမာရစ်ကို အသက်သွင်းရန်အတွက် 94 ℃ (10 မှ 15 မိနစ်) တွင် ကြာရှည်ခံရန်မလိုအပ်ပါ။PlatinumTaq DNA polymerase ဖြင့် Taq DNA polymerase လုပ်ဆောင်မှု 90% ကို 94 ဒီဂရီတွင် 2 မိနစ်အကြာတွင် ပြန်လည်ရရှိခဲ့သည်။
11. Nest-PCR
nested primers ကို အသုံးပြု၍ အသံချဲ့ထွင်မှု ဆက်တိုက်ပြုလုပ်ခြင်းသည် တိကျမှုနှင့် အာရုံခံနိုင်စွမ်းကို တိုးတက်စေနိုင်သည်။ပထမအကျော့သည် 15 မှ 20 cycles ၏ standard amplification ဖြစ်သည်။ကနဦးချဲ့ထွင်မှုထုတ်ကုန်၏ အနည်းငယ်သောအပိုင်းကို အကြိမ် 100 မှ 1000 အထိ ရောစပ်ပြီး 15 မှ 20 ပတ်ကြာ ဒုတိယအကျော့တွင် ချဲ့ထွင်ခြင်းသို့ ပေါင်းထည့်ခဲ့သည်။တနည်းအားဖြင့်၊ ကနဦး ချဲ့ထွင်ထားသော ထုတ်ကုန်ကို ဂျယ်သန့်စင်ခြင်းဖြင့် အရွယ်အစားကို တိုင်းတာနိုင်သည်။ပထမ primer အတွင်းရှိ ပစ်မှတ်အစီအစဥ်နှင့် ချိတ်ဆက်နိုင်သည့် ဒုတိယအကျော့ ချဲ့ထွင်မှုတွင် nested primer ကို အသုံးပြုသည်။nested PCR ကိုအသုံးပြုခြင်းသည် primer အစုံနှစ်ခုလုံးအတွက် ပစ်မှတ်အစီအစဥ်အနည်းငယ်ရှိသောကြောင့် ပစ်မှတ်အများအပြားကို ချဲ့ထွင်နိုင်ခြေကို လျော့နည်းစေသည်။တူညီသော primers များဖြင့်တူညီသောစုစုပေါင်းစက်ဝန်းအရေအတွက် (30 မှ 40) သည် အတိအကျမဟုတ်သောဆိုဒ်များကိုချဲ့ထွင်သည်။Nested PCR သည် ကန့်သတ်ပစ်မှတ်အစီအစဥ်များ (ဥပမာ ရှားပါး mrnas) ၏ အာရုံခံနိုင်စွမ်းကို တိုးစေပြီး ခက်ခဲသော PCRS ၏ တိကျမှုကို မြှင့်တင်ပေးသည် (ဥပမာ 5′ RACE)။
12. ဆင်းနေသော PCR
PCR ၏ပထမအနည်းငယ်စက်ဝန်းအတွက် တင်းကျပ်သောအငွေ့အသက်အခြေအနေများကိုအသုံးပြုခြင်းဖြင့် ဆင်းသက်လာသော PCR သည် တိကျမှုကိုတိုးတက်စေသည်။စက်ဝိုင်းသည် ခန့်မှန်းထားသည့် Tm ထက် 5 ℃ ပိုများသော annealing temperature တွင် စတင်သည်၊ ထို့နောက် လည်ပတ်မှုတစ်ခုစီသည် 1 ℃ မှ 2 ℃ ဖြင့် annealing temperature သည် Tm 5 ℃ အောက်တွင်ရှိသည်။အမြင့်ဆုံးသော သံယောဇဉ်ရှိသော ဦးတည်ရာပုံစံမှသာလျှင် ချဲ့ထွင်ပါမည်။ဤထုတ်ကုန်များသည် နောက်ဆက်တွဲ လည်ပတ်မှုများတွင် ဆက်လက်ချဲ့ထွင်ကာ ချဲ့ထွင်ထားသော အတိအကျမဟုတ်သော ထုတ်ကုန်များကို စုစည်းထားသည်။Descending PCR သည် AFLP DNA လက်ဗွေနှိပ်ခြင်းကဲ့သို့သော primer နှင့် target template အကြား တူညီမှုအတိုင်းအတာကို မသိနိုင်သော နည်းလမ်းများအတွက် အသုံးဝင်သည်။
ဆက်စပ် PCR Kits
2× PCR သူရဲကောင်းTMရောနှောစနစ်သည် သာမန် PCR ရောစပ်စနစ်ထက် PCR inhibitors များကို ပိုမိုခံနိုင်ရည်ရှိပြီး ရှုပ်ထွေးသော ပုံစံအမျိုးမျိုး၏ PCR ချဲ့ထွင်မှုကို အလွယ်တကူ ရင်ဆိုင်နိုင်သည်။ထူးခြားသောတုံ့ပြန်မှုစနစ်နှင့် စွမ်းဆောင်ရည်မြင့်မားသော Taq Hero သည် PCR တုံ့ပြန်မှုကို ပိုမိုချဲ့ထွင်နိုင်စွမ်း၊ တိကျမှုနှင့် အာရုံခံနိုင်စွမ်းတို့ကို ပိုမိုမြင့်မားစေသည်။
ပိုမိုမြင့်မားသောချဲ့ထွင်မှုထိရောက်မှု
၎င်းတွင် 5'→3' DNA polymerase လုပ်ဆောင်ချက်နှင့် 3'→5' exonuclease လုပ်ဆောင်ချက်မပါဘဲ 5'→3' exonuclease လုပ်ဆောင်ချက်တို့ ပါဝင်သည်။
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
တိကျသော—အကောင်းမွန်ဆုံးသော ကြားခံနှင့် hot-start Taq အင်ဇိုင်းသည် အတိအကျမဟုတ်သော ချဲ့ထွင်မှုနှင့် primer dimer ဖွဲ့စည်းခြင်းကို ဟန့်တားနိုင်သည်။
အာရုံခံနိုင်စွမ်း မြင့်မားသည်- ပုံစံပလိတ်၏ ကော်ပီ နည်းပါးမှုကို ရှာဖွေနိုင်သည်။
RT-PCR အလွယ်ᵀᴹ I (ခြေတစ်လှမ်း)
ပစ္စည်းကိရိယာသည် ထူးခြားသော Foregene ပြောင်းပြန် စာသားပြောင်းပြန်ဓါတ်ဆားနှင့် Foregene HotStar Taq DNA Polymerase နှင့် တုံ့ပြန်မှု၏ ချဲ့ထွင်မှု၏ ထိရောက်မှုနှင့် တိကျမှုတို့ကို ထိထိရောက်ရောက် မြှင့်တင်ရန် တုံ့ပြန်မှုစနစ်အား ထိထိရောက်ရောက် မြှင့်တင်ပေးပါသည်။
စာတိုက်အချိန်- မေလ-၀၉-၂၀၂၃